据统计,哺乳动物的蛋白质组包含了数百万种独特的蛋白质形态,这种蛋白质的复杂性是由于约25,000个基因的基因组、将潜在的蛋白种类增加到100,000个的转录组、以及丰富的蛋白质翻译后修饰(PTM)。尤其是PTM, 几乎所有蛋白质都会被多个PTM修饰,因此相对于转录组和基因组,经过翻译后修饰的蛋白质都大幅增加了蛋白质组的复杂性,同时也赋予了蛋白质功能多样性的无限潜力。
蛋白翻译后修饰(PTM),如磷酸化、泛素化、乙酰化、SUMO化等,在细胞信号转导、蛋白稳定和转换、蛋白识别和相互作用、空间定位等方面发挥着重要作用,具体功能如下:
| PTM | 功能 | 靶氨基酸 | 修饰物 |
| 磷酸化 | 信号转导、蛋白-蛋白相互作用、降解前体、表观遗传学 | Y, S, T | Kinase, phosphatase |
| 泛素化 | 蛋白酶体靶向-降解、蛋白-蛋白相互作用、蛋白定位、蛋白活性 | K | Ub Ligase, DUB |
| 乙酰化 | 表观遗传、代谢调节、蛋白稳定性、蛋白定位、蛋白合成 | K | HAT, HDAC |
| SUMO化 | 核蛋白定位、转录活性、蛋白稳定性、应激反应、细胞周期 | K | SAE, ubc9, SENP |
| 甲基化 | 表观遗传调控、信号转导、蛋白-蛋白相互作用 | R,K | methyltransferase,demethylase |
| 糖基化 | 蛋白折叠、蛋白结构、蛋白稳定性、细胞粘附、糖蛋白转运、可能与磷酸化竞争 S和T 的结合 | N,S,T | glycosyltransferase,glycoside hydrolase |
| 棕榈酰化 | 膜蛋白结合、蛋白的亚细胞迁移、蛋白-蛋白相互作用、蛋白稳定性 | C, S, T | palmitoyl protein thioesterases |
表1:不同类型PTM的功能和特点
多项研究表明,PTM的失调与癌症、心力衰竭、神经系统和代谢疾病等息息相关。鉴于PTM对于疾病治疗和药物开发的重要性,因此小优就给大家带来了PTM研究超全指南,内容包括:PTM在疾病研究中的综述、PTM检测方法&PTM研究工具。由于篇幅原因,小优将PTM研究超全指南分了上下部,今天我们就来详细聊聊上部——PTM在疾病研究中的综述~
1. 肿瘤中的PTM
癌症的发生是细胞通过多层调控进行重编程的一个过程,以此来满足肿瘤迁移和增殖的需要1。最近的研究揭示了控制细胞信号转导和代谢途径的重要调控层,其中PTM在其中发挥了重要的作用。此外,PTM之间的串扰以及PTM-PTM相互作用的失调已被认为与多种疾病(包括许多癌症)的发病机制有关2。
许多与癌症有关的蛋白质,包括 GPCR、p53、DAPK1、NF-kB 等,都会出现乙酰化、糖基化、磷酸化、甲基化、SUMO 化和泛素化过度表达或表达不足的情况3。因此,了解PTM在癌症中的作用为研究人员和临床医生提供了新的治疗干预点,以及用于诊断癌症、监测其进展和评估治疗效果的潜在生物标志物。
肿瘤抑制蛋白PTEN是一种磷脂酰肌醇和张力蛋白同源物,可调节细胞的黏附、迁移、增殖、生长和存活。PTEN抑制了细胞生存和生长所必需的PI3K/Akt信号通路,从而发挥对肿瘤的抑制作用4。大量的信息表明,常见的PTEN PTM(磷酸化、泛素化、SUMO化、乙酰化)会动态地改变蛋白质的稳定性、活性、定位和蛋白质间的相互作用。此外,PTEN 的翻译后调控缺陷在癌细胞中发生的频率比正常细胞要高得多,从而导致 PTEN 活性丧失,这是人类癌症中最常见的突变之一5。
同样,转录因子p53可调控多达3,000个基因的表达,这些基因涉及细胞凋亡、衰老、细胞周期停滞、DNA修复、细胞凋亡、肿瘤微环境、自噬以及肿瘤的侵袭和转移6-7。p53通过多达50个PTM进行调控,其中泛素化、磷酸化和乙酰化的调控最显著(图1) 8-9。
Wnt/β-catenin信号通路在胚胎发育过程中调节细胞的增殖、分化和迁移,并参与成年细胞的动态平衡,该信号通路是通过PTM调节的,它的失调与包括退行性疾病和癌症在内的多种疾病有关10。与大多数蛋白质一样,Wnt信号转导及其重要的分子因子受到多个PTM的调控,单个PTM能够增强和抑制其活性,这具体取决于它作用的氨基酸残基。此外,PTM的影响可能是相互协同作用的,也可能是相互排斥的。要想了解这些复杂、多样且相互影响的调控过程,从而达到诊断和治疗癌症的目的,需要用到一些灵敏的分析方法和试剂,小优将会在下一章再给大家详细介绍。
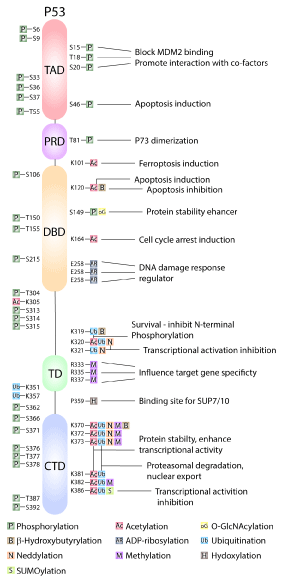 图1:p53翻译后修饰示意图
图1:p53翻译后修饰示意图
2. 心血管疾病中的 PTM
心血管疾病的病理生理学通常涉及心肌细胞的死亡和功能障碍,而心肌细胞的收缩能力取决于离子通道和泵、细胞骨架蛋白和受体的正常功能。许多相关的蛋白质都是通过PTM来调节的,PTM能够迅速并且较大地影响相关蛋白质的活性,从而影响细胞的反应11。例如,受钙控制的三聚肌钙蛋白复合体能够调节肌节的收缩,这种复合体的一个成分是心肌肌钙蛋白I(cTnI),由于会降解并出现在血液中,因此可作为心血管疾病的生物标志物12。还有研究发现了cTnI 上的其它PTM,包括乙酰化和甲基化等13,这对心肌梗塞和心力衰竭等心血管疾病的诊断和治疗具有重要意义14。
3. 干细胞研究中的PTM
多能干细胞(PSCs)包括胚胎干细胞(ESCs)和诱导多能干细胞(IPSCs),它们通过转录因子的作用来维持原始分化状态、激活多功能促进基因,同时抑制分化促进基因。这些转录因子的表达水平和转录激活能力受PTM调节的调控15。
转录因子Oct4,Sox2和Nanog通过复杂的机制调控转录,其中每个转录因子都能独立发挥作用,也能够自我抑制16,这三种转录因子都通过PTM进行调控。Oct4的表达水平和转录活性受泛素化、SUMO化和磷酸化的调控,其中泛素化的Oct4降低了转录水平并诱导ESC分化17。Nanog在多个丝氨酸残基上发生磷酸化,从而降低其活性并增强 ESC 分化18,而Sox2 在 ESC 中的表达是通过甲基化和磷酸化之间的竞争来调节的19。
4. PTM和药物开发
PTM通过调节蛋白质的物理和化学性质、折叠、构象、稳定性和活性来影响细胞发育过程,从而影响多种疾病的病因。PTM可能发生的位置、数量以及翻译后变化的多样性,将起源于大约21,000个基因的蛋白质组转化为具有独特功能和活性的数百万个蛋白质20-21。我们对PTM在疾病中发挥作用的了解仍然主要集中在PTM的局部效应、它们与药物结合位点以及它们参与蛋白质相互作用过程22。然而,面临这些困难和挑战,从事药物开发的研究人员都一一克服,并且设计出了能增强或破坏PTM以及携带PTM的蛋白质活性的蛋白质药物和小分子药物。
相关产品推荐
| 货号 | 品名 | 应用 |
| abs130624-50ug | Rabbit anti-Phospho-NF-kappaB p65(Ser536) Polyclonal Antibody | 磷酸化抗体 |
| BK160 | Signal-Seeker™ Phosphotyrosine Detection Kit (30 assays) | 磷酸化检测试剂盒 |
| AAC02-Beads | Acetyl-Lysine Affinity Beads-AAC02 | 乙酰化磁珠 |
| BK163-S(10assays) | Signal-Seeker™ Acetyl-Lysine Detection Kit (10 assays) | 乙酰化检测试剂盒 |
| BML-UW0155-0025 | Ubiquitin vinyl sulfone, (HA-tag) | 泛素化探针 |
| BK161 | Signal-Seeker™ Ubiquitin Detection Kit (30 assays) | 泛素化检测试剂盒 |
| BML-UW9195-0500 | SUMO-1 (human), (recombinant) (His-tag) | SUMO化蛋白 |
| BK162 | Signal-Seeker™ SUMOylation Detection Kit (30 assays) | SUMO化检测试剂盒 |
参考文献:
1. Martin-Bernabe A. et al. 2017. The importance of post-translational modifications in systems biology approaches to identify therapeutic targets in cancer metabolism. Curr. Opin. Struct. Biol. 3, 161–169.
2. Zheng W. et al. 2019. Crosstalk of intracellular post-translational modifications in cancer. Arch. Biochem. Biophys. 676, 108-138.
3. Sharma B.S. et al. 2019. Post-translational modifications (PTMs), from a Cancer Perspective: An Overview. Oncogene. 2. 10.35702/onc.10012.
4. Song M.S. et al. 2012. The functions and regulation of the PTEN tumor suppressor. Nat. Rev. Mol. Cell Biol. 13, 283–296.
5. Chalhoub N. and Baker S.J. 2009. PTEN and the P13-Kinase Pathway in Cancer. Annu Rev Pathol. 4, 127–150.
6. Bieging K.T. et al. 2014. Unravelling mechanisms of p53-mediated tumour suppression. Nat. Rev. Cancer. 14, 359–370.
7. Kruiswijk F. et al. 2015. p53 in survival, death and metabolic health: a lifeguard with a licence to kill. Nat. Rev. Mol. Cell Biol. 16, 393–405.
8. Kruse J.P. and Gu W. 2009. Modes of p53 regulation. Cell. 137, 609–622.
9. Bode A.M. and Dong Z. 2004. Post-translational modification of p53 in tumorigenesis. Nat. Rev. Cancer. 4, 793–805.
10. Polakis P. 2000. Wnt signaling and cancer. Genes Dev. 14, 1837–1851.
11. Liddy K. et al. 2013. Functional decorations: post-translational modifications and heart disease delineated by targeted proteomics. Genome Med. 5, 20.
12. Wijnker P.J.M. et al. 2014. Troponin I phosphorylation in human myocardium in health and disease. Neth. Heart J.22, 463–469.
13. Zabrouskov V. et al. 2008. Unraveling molecular complexity of phosphorylated human cardiac troponin I by top down electron capture dissociation/electron transfer dissociation mass spectrometry. Mol. Cell Proteomics. 7, 1838–1849.
14. Onwuli D.O. et al. 2019. The inhibitory subunit of cardiac troponin (cTnI) is modified by arginine methylation in the human heart. Int J Cardiol. 282, 76–80.
15. de los Angeles A. et al. 2015. Hallmarks of pluripotency. Nature. 525, 469–478.
16. Pan G. et al. 2006. A negative feedback loop of transcription factors that controls stem cell pluripotency and self-renewal. FASEB J. 20, 1730–1732.
17. Xu H.M. et al. 2004. Wwp2, an E3 ubiquitin ligase that targets transcription factor Oct-4 for ubiquitination. J. Biol. Chem. 279, 23,495–23,503.
18. Kim S.H. et al. 2014. ERK1 phosphorylates Nanog to regulate protein stability and stem cell self-renewal. Stem Cell Res. 13, 1–11.
19. Fang L. et al. 2014. A methylation-phosphorylation switch determines Sox2 stability and function in ESC maintenance or differentiation. Mol Cell. 255, 537–551.
20. van Kasteren S.I. et al. 2007. Expanding the diversity of chemical protein modification allows post-translational mimicry. Nature. 446(7139).1105–1109.
21. Lee T.Y. et al. 2006. dbPTM: an information repository of protein post-translational modification. Nucleic Acids Res. 34(Database issue), D622–D627.
22. Min-Gang S. et al. 2017. Investigation and identification of functional post-translational modification sites associated with drug binding and protein-protein
interactions. BMC Syst Biol. 11 (Suppl7), 132.









 危险品化学品经营许可证(不带存储) 许可证编号:沪(杨)应急管危经许[2022]202944(QY)
危险品化学品经营许可证(不带存储) 许可证编号:沪(杨)应急管危经许[2022]202944(QY)  营业执照(三证合一)
营业执照(三证合一)