引言
自上世纪90年代以来,基于蛋白质结构的药物设计(Structure-Based Drug Design, SBDD)一直是创新药物发现的主流方法。SBDD通过解析蛋白质的三维结构,识别潜在的配体结合位点,并通过虚拟筛选或全新设计发现活性化合物,在针对具有明确靶标的疾病治疗方面取得了重大进步。然而,SBDD方法也面临诸多挑战,如许多蛋白质并没有高分辨率的结构,精确预测配体结合口袋困难,以及虚拟筛选过程中可能出现的假阳性问题。

为了解决这些挑战,中国科学院上海药物研究所郑明月团队提出了一种基于序列的药物设计新方法,并在Nature Communications期刊上发表了题为“Sequence-based drug design as a concept in computational drug design”的研究论文。这一新方法采用端到端的可微学习,直接从蛋白质序列出发进行药物设计,避免了SBDD中的多个复杂步骤,为创新药物发现提供了新的视角。
一、基于蛋白质结构的药物设计(SBDD)的挑战
SBDD方法的核心在于解析蛋白质的三维结构,进而识别配体结合位点,通过虚拟筛选或全新设计发现活性化合物。然而,SBDD方法在实际应用中面临多重挑战:
-
蛋白质结构获取困难:许多蛋白质并没有高分辨率的结构信息,尤其是膜蛋白、大分子复合物等复杂蛋白质结构更是难以获得。
-
配体结合口袋预测困难:配体结合口袋往往位于蛋白质的表面凹陷处,其形状和大小各异,且往往违反“蛋白质折叠规则”。此外,配体的结合会诱导氨基酸构象发生变化,进一步增加了预测的难度。
-
变构效应多样性:对于具有多个结构域的新靶标,如何确定配体结合位点是一个难题。不同结构域之间的相互作用和变构效应增加了靶标识别和药物设计的复杂性。
-
虚拟筛选假阳性问题:虚拟筛选过程中可能会产生大量的假阳性结果,误差和错误的不断累积会导致严重的假阳性问题,增加了后续实验验证的难度和成本。
二、基于序列的药物设计新方法
为了克服SBDD方法的局限性,郑明月团队提出了一种基于序列的药物设计新方法。这一新方法采用端到端的可微学习,直接从蛋白质序列出发进行药物设计,避免了SBDD中的多个复杂步骤。
- 端到端的可微学习
端到端的可微学习已经在计算机视觉和自然语言处理等领域产生了革命性的影响。这一概念的核心在于用可微基元(differentiable primitives)取代复杂流程的所有组件,从输入端到输出端进行联合优化。AlphaFold2在蛋白质结构预测方面的成功很大程度上依赖于端到端可微学习的思想。
在药物设计领域,端到端的可微学习可以以一种自洽和数据高效的方式执行整个学习建模过程,避免复杂流程中的错误积累。郑明月团队设计的Transformer CPI 2.0模型正是基于这一思想,利用Transformer神经网络架构和预训练蛋白质语言模型,在蛋白质和化学多样性空间中都展示出了良好的泛化能力。
- Transformer CPI 2.0模型
Transformer CPI 2.0模型是郑明月团队提出的一种基于序列的药物设计新方法的核心工具。该模型利用Transformer神经网络架构和预训练蛋白质语言模型,从蛋白质序列信息中有效学习蛋白质与配体分子间相互作用的抽象知识。
Transformer CPI 2.0模型的架构包括输入层、Transformer编码器和输出层。输入层将蛋白质序列和化合物结构信息转化为特征向量;Transformer编码器通过自注意力机制和位置编码对输入特征进行编码;输出层则输出预测的蛋白质-配体相互作用得分。
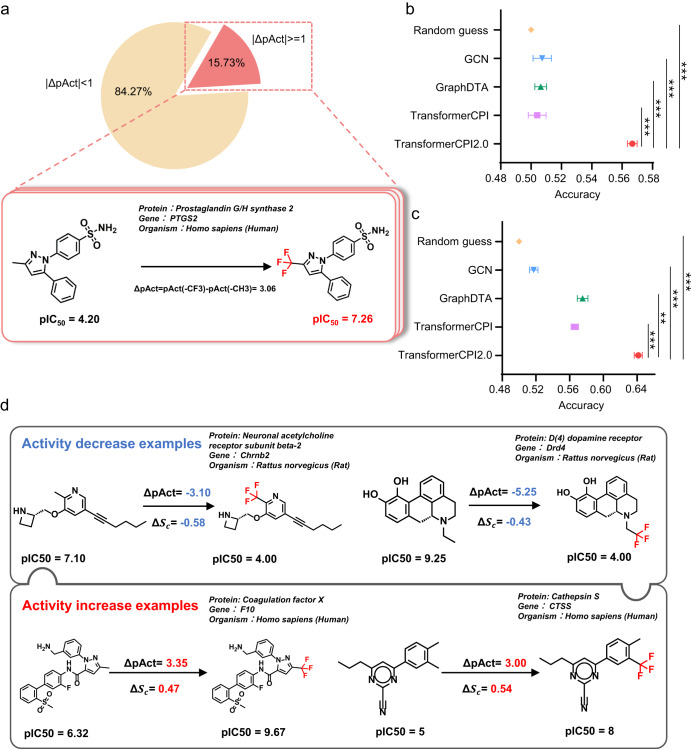
通过对Transformer CPI 2.0模型进行可解释分析,结果表明模型可以通过注意力机制从序列信息中有效学习蛋白质与配体分子间相互作用的抽象知识,而不仅是简单的记忆训练数据中的分布偏差。这一特性使得Transformer CPI 2.0模型能够推广到新的蛋白质和化学空间,显示出良好的泛化能力。
- 概念验证与实验结果
为了进一步检验基于蛋白质序列药物设计的可行性,郑明月团队基于Transformer CPI 2.0预测结果开展了湿实验的验证研究。研究团队针对多种具有挑战性的药物靶标成功发现活性化合物,包括靶向E3泛素连接酶接头蛋白SPOP(配体结合位点为难以靶向的PPI作用界面)的新骨架抑制剂和靶向E3泛素连接酶RNF130(无蛋白晶体结构、无已知活性化合物)的结合分子。
此外,通过逆向应用Transformer CPI 2.0对已上市药物进行蛋白质组范围的靶标筛选,成功发现雷贝拉唑抗肿瘤作用的潜在靶标ADP-核糖基化因子ARF1。这些新发现的活性分子和靶标蛋白都是模型训练过程未见的,表明Transformer CPI 2.0可以推广到新的蛋白质和化学空间,显示了良好的泛化能力。
三、基于序列的药物设计方法的优势与应用前景
基于序列的药物设计方法相比传统的SBDD方法具有诸多优势,并在创新药物发现领域展现出广阔的应用前景。
- 无需高分辨率蛋白质结构
基于序列的药物设计方法直接从蛋白质序列出发进行药物设计,无需高分辨率的蛋白质结构信息。这一特性使得该方法在无法获得高质量蛋白质3D结构的场景中发挥作用,为创新药物发现提供了新的视角。
- 泛化能力强
基于序列的药物设计方法通过端到端的可微学习,从输入端到输出端进行联合优化,避免了复杂流程中的错误积累。这使得该方法具有良好的泛化能力,能够推广到新的蛋白质和化学空间,发现新的活性分子和靶标蛋白。
- 加速药物发现进程
基于序列的药物设计方法可以与其他虚拟筛选和高通量体外筛选技术结合,加速药物发现进程。通过计算机、信息学、化学和生物学等多学科融合交叉,该方法为创新药物发现提供了高效、精准的工具。
- 超大规模化合物库筛选
随着超大规模按需定制化合物库(make-on-demand library)技术的快速发展,已覆盖了数亿到数十亿种化学物质的多样性空间。基于序列的药物设计方法可以有效地简化建模和筛选流程,更加高效地探索未知化学和生物学空间,为原创药物的发现提供新的出发点和切入点。
四、结论与展望
郑明月团队在Nature Communications期刊上发表的研究论文,提出了一种基于序列的药物设计新方法,为创新药物发现提供了新的视角。该方法采用端到端的可微学习,直接从蛋白质序列出发进行药物设计,避免了SBDD中的多个复杂步骤,显示出良好的泛化能力和应用前景。
未来,基于序列的药物设计方法有望在以下方面取得进一步突破:
-
提高预测精度:通过优化模型架构和训练策略,进一步提高基于序列的药物设计方法的预测精度,为创新药物发现提供更加可靠的工具。
-
拓展应用范围:将基于序列的药物设计方法应用于更多类型的药物靶标和疾病领域,发现更多具有临床价值的创新药物。
-
结合其他技术:与其他虚拟筛选和高通量体外筛选技术结合,形成更加完善的药物发现流程,加速创新药物的研发进程。
-
推动跨学科合作:加强计算机、信息学、化学和生物学等多学科的交叉合作,推动基于序列的药物设计方法的不断创新和发展。
| 名称 | 货号 | 规格 |
| DYKDDDDK Tag (D6W5B) Rabbit mAb (Binds to same epitope as Sigma-Aldrich Anti-FLAG M2 antibody) | 14793S | 100ul |
| beta-Tubulin (D2N5G) Rabbit mAb | 15115S | 100ul |
| Phospho-Akt (Thr308) (244F9) Rabbit mAb | 4056S | 100ul |
| PTEN (138G6) Rabbit mAb | 9559L | 300ul |












 危险品化学品经营许可证(不带存储) 许可证编号:沪(杨)应急管危经许[2022]202944(QY)
危险品化学品经营许可证(不带存储) 许可证编号:沪(杨)应急管危经许[2022]202944(QY)  营业执照(三证合一)
营业执照(三证合一)