文章描述一种靶向转录组学方法结合表面蛋白测量,以捕获免疫细胞在低测序的异质性深度,直观地探索单细胞水平上蛋白质和转录表达的关系。
△点击放大图片
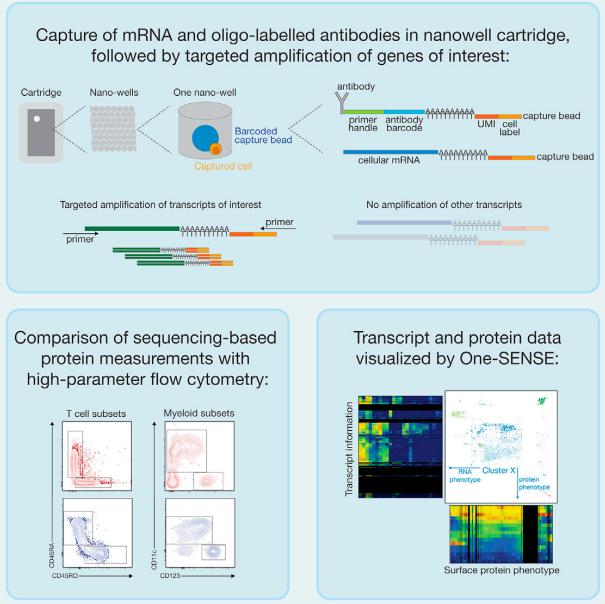
高通量单细胞RNA测序(scRNAseq)已成为评估免疫细胞异质性的常用工具。获取蛋白质表达数据和转录组数据解决了仅评估转录本固有的一些限制,但也使每个单个细胞所需的测序读深度几乎两倍。
这里描述了一种靶向转录组学方法,该方法结合了超过400个基因的分析和同时测量2 X104个细胞上的超过40个蛋白质。与全转录组方法相比,这种靶向方法只需要大约十分之一的读取深度,同时对低丰度转录本保持高敏感度。
#1 实验方法:
T细胞刺激试验
使用磁激活细胞分选(MACS)将PBMCs(Seattle Area Control Cohort)去除新鲜解冻的髓系细胞、B细胞和表达IL3Ra的细胞;
MACS抗体针对CD33、CD20和CD123和抗小鼠IgG微珠(骨髓细胞#130-048-402);纯化细胞部分用10%胎牛血清和PMA(10ng/mL)和离子霉素(1mg/mL)在37℃下洗涤和刺激60分钟;
未受刺激的细胞无PMA/离子霉素在37℃孵育60分钟;
之后,细胞被靶向CD3、TCRab、CD4、CD8、PD1的抗体结合物CD137、CD103、CD69、CD39、CCR7、CD45RA、CD45RO和Tim3;
并接受下面描述的目标转录组工作流程,使用BD Rhapsody™ T Cell Expression Panel HS。
流式细胞术
对于流式细胞术分析,解冻后,PBMCs与Fc阻断试剂和可固定的紫外蓝活/死试剂在PBS中孵育15分钟;
如果需要,细胞用EBV-四聚体试剂在流式细胞仪缓冲液(2%胎牛血清PBS)中稀释30分钟,然后室温洗涤两次;
然后,细胞在室温下与100mL总体积的抗体主混合液中新鲜制备,孵育20分钟;
然后进行两次洗涤;
所有抗体均被滴定并进行最佳稀释,染色程序在96孔圆底板中进行;
表S4列出了使用面板的详细列表,包括荧光染料和所有抗体的最终稀释度;
染色细胞用4%PFA室温固定20分钟,洗涤,在FACS缓冲液中重悬,在4℃黑暗中保存直到上机;
上机
所有样品均采用FACSymmioA5采集,配备30个探测器和355nm、405nm、488nm、532nm和628nm激光器和FACSDiva。使用改进的电压滴定方法优化探测器增益,并每天使用6峰超彩虹珠进行标准化。每次实验均使用FACS缓冲液或死活染料稀释的抗体捕获珠制备单染色对照。
数据分析
采集后,数据以FCS3.1格式导出,并使用FlowJo进行分析。FSC-A和FSC-H门控排除了粘连体。对于其中一些图,使用适当的FlowJo插件对获得的单元格数量进行降采样,以匹配AbSeq工作流中分析的单元格数量。
细胞分选
所有细胞分选均在FACSAriaIII上进行,配备20个探测器和405nm、488nm、532nm和628nm激光器。
细胞被分选到含有500mL完全RPMI的冷冻Eppendorf管中,用PBS洗涤一次,并立即用于后续处理。
靶向转录组和蛋白单细胞文库的制备和测序
对于所有的靶向转录组学实验,我们使用了BD Rhapsody单细胞分析系统。
1.CD45+活PBMCs和EBV-四聚体+CD8+T细胞依次使用BD™ Hu Single Cell Sample Multiplexing Kit和BDAB-AbSeq试剂(BDOligos试剂进行序列标记。简单地说,来自每个供体或亚型细胞的细胞(分类后)用样本标记。
2.每个样品用FACS缓冲液洗涤两次,然后将所有样品汇集在一起。
3.汇集的样品再次清洗一次,然后用200mL的FACS缓冲液中稀释的AbSeqAb-Oligos染色(如表S4所示(图文最后))。
4.然后将汇集的样品洗涤两次,计数并在冷BD样品缓冲液中重悬,在620mL中获得约20,000个细胞。
5.使用单细胞捕获和BD Rhapsody单细胞分析系统从合并的样本中分离单细胞。
最终文库的质量使用安捷伦2200高灵敏度D5000屏幕磁带进行评估,并使用量子位dsDNAHS试剂盒使用量子位荧光仪进行定量。
6.将最终的文库稀释到2nM,并在HiSeq2500测序仪(Illumina)上进行多端(150bp)测序。
最后进行全转录组单细胞文库的制备和测序。
#2 寡核苷酸标记的抗体探针与高维流式细胞术的比较
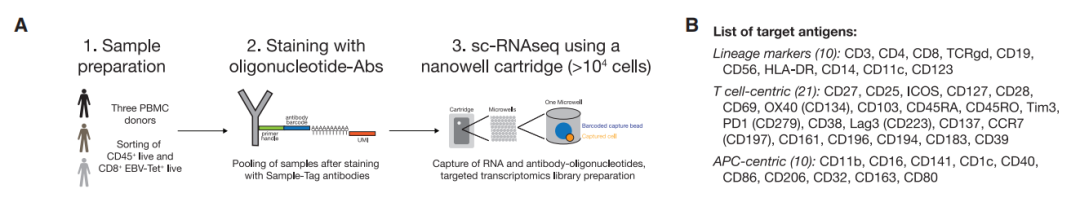
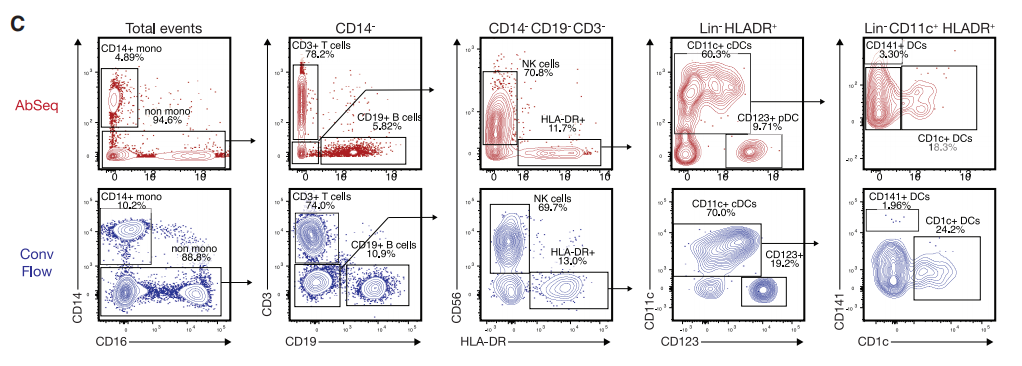


图1
△点击放大图片
(A).描述实验工作流程的示意图。来自三个供体的PBMC样本(携带HLA-A*02:01等位基因的健康对照)被分成两半,一个等分用于多组学工作流程,一个等分用于使用两个30个参数面板的流式细胞仪表型。该等位基因允许使用EBV四聚体试剂分离EBV特异性CD8+T细胞。为了确保这些罕见的抗原特异性T细胞的足够数量,通过荧光激活细胞分选(FACS)富集四聚体阳性T细胞。同时,从PBMCs中分类了CD45+活的白细胞。
(B).在多组学和常规流式细胞术实验中使用的抗体靶点的概述。
(C).使用结合的AbSeq数据集(红色)和从传统(conv)流式细胞术数据集(蓝色)获得的连接和下采样事件(27000个细胞)对主要免疫亚群进行手动门控:髓系。即使低丰度的细胞群,如CD1c+常规树突状细胞(cDCs)和交叉呈现的CD141+cDCs,也可以通过其表面蛋白表型清楚地识别出来。
(D).通过使用组合的AbSeq数据集(红色)和从细胞术数据集(蓝色)中连接的下采样事件,手动对各种T细胞标记物进行门控:淋巴系。
(E)和(F).在两个不同的数据集中,所有种群都以相当的频率出现,在包括细胞分选在内的实验中,髓系细胞的丰度略低。
#3 目标转录组学捕获类似于全转录组方法


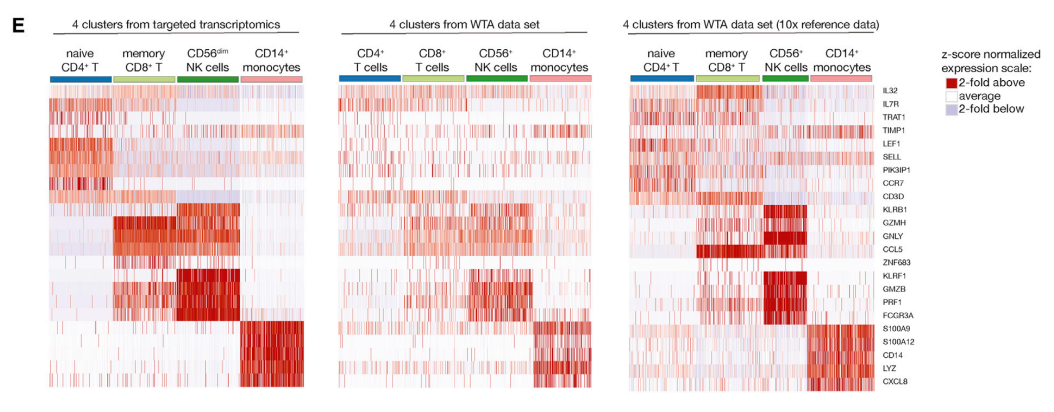
图2
△点击放大图片
(A).来自一个代表性供体(8843个细胞)的转录数据聚类显示在UMAP(均匀流形近似投影)图上。集群已经通过关键谱系基因的表达进行了注释。
(B).使用Seurat实现的MAST(基于模型的单细胞转录组分析)识别每个聚类的前10个差异表达基因,并在Z评分归一化后在热图上可视化。集群名称采用与(A)中相同的配色方案显示。CD4+T细胞和CD8+T细胞分离得很好,我们观察到表达FOXP3和CTLA4的Treg作为一个单独的簇。
(C).在三个不同的CD4+T细胞簇上的指示转录本和蛋白的表达,突出了CD25+CD127低Treg簇(橙色)。
(D).所有检测到的转录本相对于来自同一供体的全转录组数据集的相对检测率。根据基因的相对检测率,人工将基因分为四组。
(E).4个具有代表性的主要免疫群体的表达模式,在目标转录组数据集上的全转录组数据(左)、来自同一供体的全转录组数据(中间)和公开可用的全转录组参考数据集(右)。
可视化热图上这四个簇的顶级差异表达基因产生了非常相似的模式,这表明具有免疫聚焦引物面板的靶向转录组学在单细胞水平上忠实地再现了PBMCs的细胞异质性。
#4 多组学靶向转录组学识别典型的记忆T细胞群,研究罕见抗原特异性CD8+T细胞

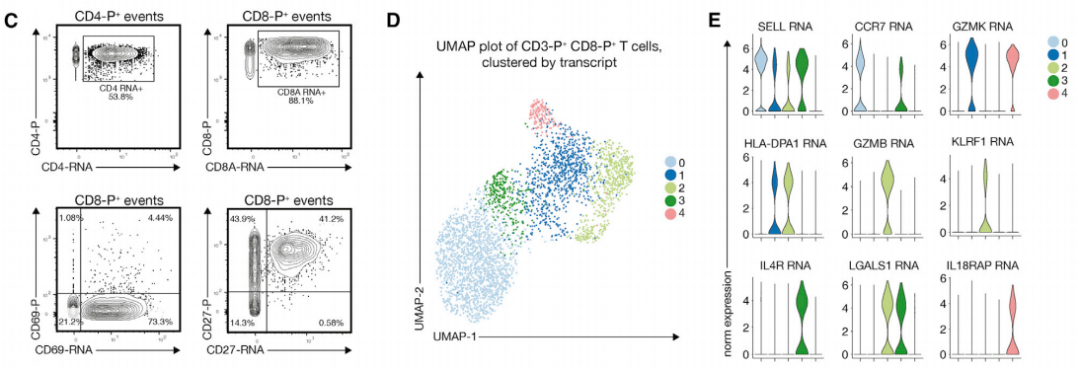
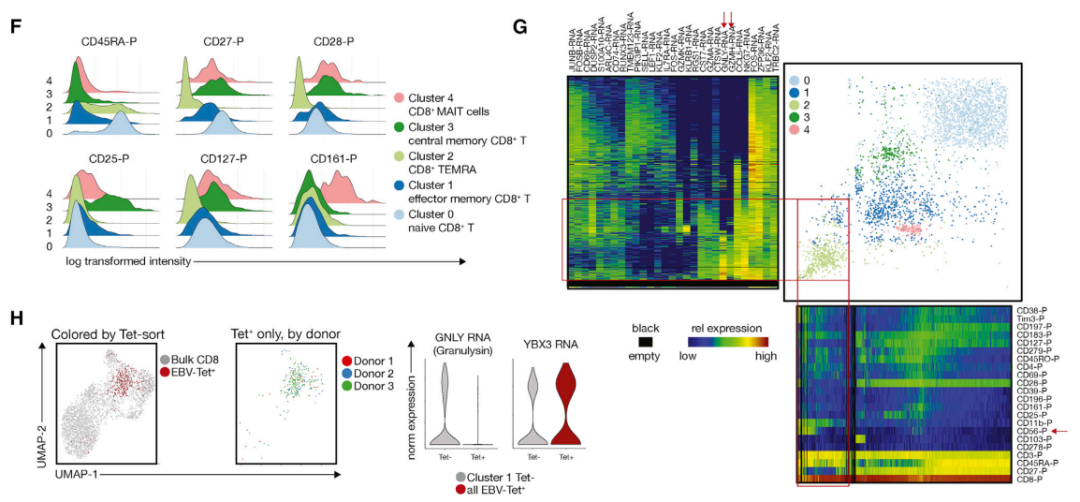
图3
△点击放大图片
(A).在单独的UMAP图上可视化了从三项专利的PBMCs的总CD45+活细胞中收集的蛋白质和RNA数据。发现,来自不同供体的细胞根据细胞类型而不是供体而聚集和分离,这表明没有由单个供体驱动的簇(左),此外,在使用转录本或蛋白质计算的UMAP图上,这两个实验批次没有显示出明显的分离(右)。
(B).示例UMAP图代表了主要免疫谱系蛋白标记物的表达,这允许明确识别CD4+和CD8+T细胞、CD19+B细胞、CD14+以及CD16+髓系细胞。
(C).显示CD4和CD69的转录本和蛋白水平的相关性较差,CD8和CD27的相关性较好。正如预期的那样,外周血中只有少数CD8+T细胞在其表面表达CD69蛋白,是在y轴上绘制的;在大量的CD8+T细胞中检测到CD69-RNA,是在x轴上绘制的。
(D).我们继续使用scamp、分析CD8+T细胞。使用转录本信息的无偏图聚类表明存在5个不同的细胞簇。
(E).对顶级差异表达基因,如SELL(编码CD62L)、CCR7和GZMB的可视化表明,这5个簇反映了典型的幼稚和记忆T细胞群体。此外,我们的数据允许识别CD8+粘膜相关不变T(MAIT)细胞,这些细胞表达高水平的白细胞介素-18受体辅助蛋白(IL18RAP)和肿瘤坏死因子
(F).我们通过表面蛋白表达证实了这些群体的相似性(图3F),中央记忆CD8+T细胞表达低水平的CD45RA蛋白和高水平的CD27和CD28蛋白,重要的是,剪接变体CD45RO和CD45RA不能通过单独分析转录本来区分,这突出了结合蛋白和转录本分析的附加价值。
(G).不同程度的一致性和能力,以区分细胞亚群从基因和蛋白质表达谱。热图中的黑色区域是指到单感图中没有细胞的区域。这种方法可以很容易地识别与转录本相似但被蛋白质分离的细胞簇,反之亦然。图(红色框和箭头)突出显示了一个例子,其中簇2(浅绿色,TEMRA细胞)通过转录本相对同质,但可以通过CD56蛋白表达分离。簇1(暗蓝色,效应记忆CD8+T细胞)和2(浅绿色,TEMRA)之间的部分细胞具有相同的蛋白特征,但可以通过GNLY和GZMH的表达来区分(红框和箭头)。
(H).三个供体的EBV特异性T细胞具有显著的相似性,大多数细胞聚集在效应记忆CD8+T细胞簇中。为了确定靶向转录组学是否适合于研究罕见抗原特异性T细胞群,我们分析了识别HLA-A*02限制性EBV表位yvldhlivv。
#5 PMA和离子霉素刺激1小时后T和NK细胞室的多组学分析

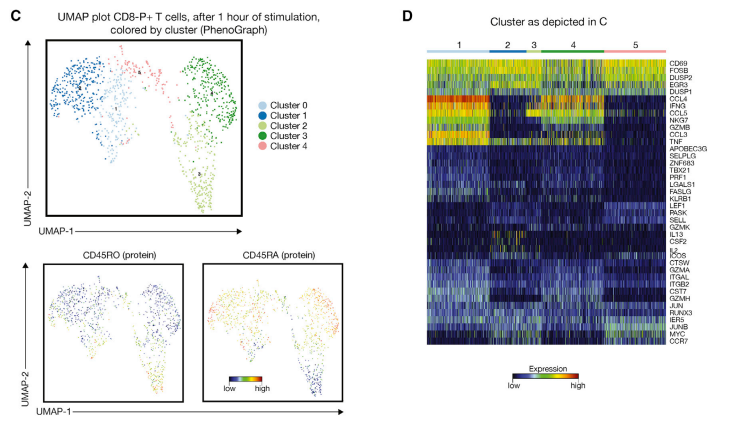
图4
△点击放大图片
(A).代表性图显示,刺激后(红色)相对于未受刺激的细胞,选定的效应转录本,如IFNG、FASL和ICOS的上调(蓝色)。
(B).在CD8蛋白+T细胞中,早期激活标记物CD69和IFNG和TNF转录本的表面蛋白表达断开。蓝色的覆盖层表示未受刺激的细胞,红色表示受刺激的细胞。
(C).受刺激的CD8蛋白+T细胞的UMAP图显示5个表型定义的簇和相应的CD45RA和CD45RO蛋白表达。
(D).显示(C)中确定的集群中关键效应转录本表达的热图,这五个簇主要由CCL3、CCL4、IFNG、TNF和各种颗粒酶的差异表达来定义。
T细胞和NK细胞的短期刺激显示了趋化因子的异质性,并与早期激活标志物CD69脱节。总的来说,该分析显示了CD8+T细胞室内相当大的功能多样性,在刺激1小时后可检测到。
#6 外周髓系细胞室的结合蛋白和转录本表型显示表面蛋白表型未捕获的炎症亚群

图5
△点击放大图片
(A).UMAP图和外周非t/非NK/非b细胞室的基于图的聚类,揭示了5个不同的群体。
(B).CD14和CD16蛋白的表达明显分离。
(C).在(A)中突出显示的5个聚类中,通过MAST识别出的顶级差异表达基因的热图。
(D).这5个簇的蛋白特征可以识别出典型的CD123+浆细胞样树突状细胞、CD1c+常规树突状细胞和CD16+单核细胞,但其中两个簇定位于CD14+单核细胞。
(E).使用One-sense进一步探索簇0和1之间的关系,揭示了这两个群体在表面蛋白剖面(CD14+CD16经典单核细胞)方面非常相似,但被一组编码促炎细胞因子和趋化因子的特定转录本分开。
(F).小提琴图显示了各自髓系群体的关键基因(上图)和簇0和簇1之间的差异表达基因,表明在CD14+CD16单核细胞中存在一个炎症亚群,表达高水平的IL1B、TNF、CXCL3和CCL4。
结合蛋白质和转录组数据,我们可以观察到外周CD14+髓系群体中的多个功能亚群,而这些亚群仅通过表面标记物表达并不明显。总之,这些数据强调了靶向转录组学可以用于不同免疫隔间的探索性研究。
为了充分利用多组学单细胞分析方法的优势,必须收集大量的细胞,以充分代表低丰度的细胞群,如抗原特异性T细胞或抗原提呈细胞(APCs)。这可以用DC亚群来说明;CD1c+cDCs通常只占外周血细胞的0.1%-0.5%,也就是说,要捕获50个不需要事先富集的细胞,需要对至少10,000个细胞进行测序。然而,如果在这样一个罕见的群体中存在明显的异质性,即使是50个细胞也可能不足以推断功能潜力。
这可以用DC亚群来说明;CD1c+cDCs通常只占外周血细胞的0.1%-0.5%,也就是说,要捕获50个不需要事先富集的细胞,需要对至少10,000个细胞进行测序。然而,如果在这样一个罕见的群体中存在明显的异质性,即使是50个细胞也可能不足以推断功能潜力。
这里描述的靶向转录组学方法提供了一个替代平台,它显著降低了转录本测序饱和所需的reads数量,但仍然提供了492个免疫中心基因的有价值的信息。
文中采用了一种成功用于高维细胞术数据的分析方法,通过可视化一维上相对于测量的蛋白质表型的顶级差异表达基因,该方法允许轻松地剖析在转录表型上相似但在蛋白质表型上不同的细胞。
表S4
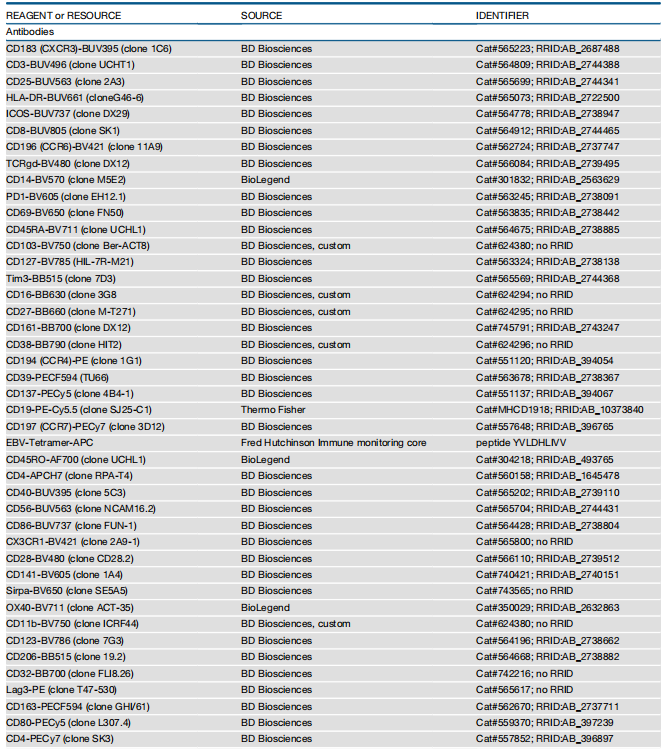


△点击放大图片
Reference:A Targeted Multi-omic Analysis Approach Measures Protein Expression and Low-Abundance Transcripts on the Single-Cell Level.