核酸酶靶向切割和释放 (CUT&RUN),与染色质免疫沉淀 (ChIP) 检测一样, 是一项用于在细胞天然染色质环境下检测蛋白-DNA 相互作用的强大通用技术。该技术自2020年问世以来,以所需细胞量少、背景低、易操作等多种优势,被广大科研工作者所青睐,也收到了来自终端用户的操作细节问题及经验,今天天特别共享给大家。
本文特别感谢中国医学科学院血液学研究所王建祥、王敏团队刘钰同学整理问题,CST技术专家详细解答。
PART 1 CUT&RUN实验基本步骤
以CST 备受欢迎的CUT&RUN试剂盒#86652为例:首先将细胞固定在伴刀豆球蛋白A磁珠上,然后用洋地黄皂苷通透细胞膜,加入一抗和pAG-MNase(Protein A-Protein G-微球菌核酸酶),一抗募集pAG-MNase到靶蛋白上,0度条件下添加Ca2+激活pAG-MNase,并切割靶蛋白两侧的DNA,使其从基因组中释放出来,扩散到上清中。上清中的DNA纯化后进行qPCR或NG-Seq鉴定、定量。
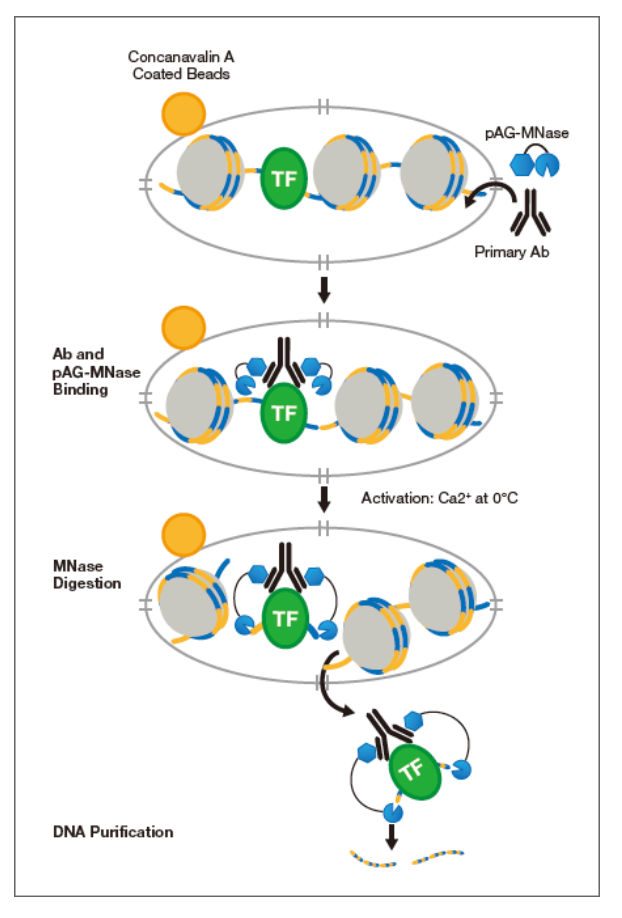
△点击放大图片
PART 2 CUT&RUN细节灵魂问答
1.Input样本问题
问:对每组的3-4个生物学重复样本,需要分别设置input对照吗,必须要和目的抗体组同样的细胞数吗?因为所用原代细胞比较珍贵,如果这样的话就要拿出相当一部分的细胞来做input对照?
答:生物学重复不需要,不同处理组需要。Input需同样的细胞数。
问:Input没有切出来100-600bp大小,是超声的问题吗?还是加错试剂了?
答:有可能超声不够,DNA没有释放出来;或者纯化后丢失;或者qPCR的过程有问题。片段很大的话无法被柱子回收,要用酚氯仿抽提。超声建议接触式超声(官网步骤),使用设置为 6 且配有 1/8 英寸探针的 VirTis Virsonic 100 超声波均质器/超声波仪以 5 组 15 秒脉冲进行超声处理,可充分碎裂input染色质。(测试不同的超声仪功率设置和/或超声处理持续时间来凭经验确定)。
问:Input这里染色质打断的原理是什么呢,DNA extraction buffer会有破膜的功能吗,不然只有DNA extraction buffer再加一个超声真的能打断吗?
答:DNA extraction buffer是有有破膜的功能的,input样品的片段化,就是在buffer里超声。
问:Input样本超声后有泡沫、温度有些高有影响吗?还是说只要跑胶后片段大小在100-600bp即可呢?
答:无影响,超声打断染色质即可(目的不是收蛋白)尽量避免,可以下调amplification。
问:后续用于建库的input量一般选择多少呢?5ng?
答:input提供的是样本中DNA(未经cut&run)的整体信息,足够建库即可,5ng是足够的。Input量较多,有5ng即可。
问:超声后误吸到沉淀会对后续的结果有影响吗?
答:尽量不要吸到沉淀。如果吸到了,就再离心一次,不要吸到沉淀。沉淀可能影响ligation。
2.活细胞制备:
问:温热200X 蛋白酶抑制剂混合物 #7012 和 100X 亚精胺 #27287,室温还是37℃?融化后如何保存,是室温放着?
答:是的,-20℃取出后置于室温,可轻弹管子促进溶解。待完全溶解混合均匀后再取用。完成后建议冻回-20℃,短时可置于冰上/4度冰箱。
问:活细胞制备:洗涤缓冲液总体积如何配制?input里面不需要蛋白酶抑制剂混合物和亚精胺,为什么?亚精胺作用是什么?
答:洗涤缓冲液总体积计算:每个细胞系(或者样本) 2 ml,每个反应或每份input样品额外准备 100 µl,比如有一个样本,有阴性对照、阳性对照、实验组三个反应+一个input,就需要2.4ml 洗涤缓冲液。input的目的是提全部DNA,对DNA和蛋白相互作用的保护并不Care,所以可以不加蛋白酶抑制剂混合物和亚精胺的。亚精胺作用是对DNA和蛋白相互作用的保护。替代了Mg离子,在正常CR实验后续需要有Mg, 来帮助内切,因为Mg会影响DNA稳定性,所以用这个替代。
问:细胞准备完毕后可以在4℃放置一段时间吗?还是要立即进行ConA珠子和一抗的结合呢?
答:不能4℃,只能常温。活细胞就先做conA活化,放冰上等样本制备好,尽量不要让活细胞stress(珠子等样本,而非样本等珠子)。
3.ConA beads和一抗的结合:
问:ConA beads用枪吹匀就可以吗?
答:是的,轻柔混匀,不要震荡。
问:ConA beads提前制备后放在冰上时间久了会有结块或者磁珠析出,用移液器吹打后可混匀,请问这样有影响吗?
答:没有影响。跟样本混合前,不是析出,只是珠子的沉降。
问:Digitonin buffer取出后在100℃放置10min后仍可以看到白色悬浮颗粒,请问这样是可以用的吗?还是需要延长100℃时间呢?放到冰上后可以看到有白色沉淀在底部,是可以直接用的吗,还是要怎样使其溶解呢?
答:延长时间溶解,时不时晃,必须保证digitonin完全溶解后再使用,否则会影响破膜效率及后续抗体,MNase切割和染色质的充分释放。可通过适当延长孵育时间保证其完全溶解。溶解后放4℃。
问:在整个的实验过程中,比如4℃ 1h等,期间Digitonin buffer可以一直放置在冰上吗?还是要放回-20℃再重新解冻呢?
答:一天protocol,可放4℃。两天放-20℃。
问:4℃孵育一抗过程中,在旋转后会有beads析出或者挂到侧壁的情况,用移液器吹打后可混匀,这种是可以的吗?一抗孵育的时间2h和过夜哪个更好呢?一抗时间越长越好吗?
答:珠子结块是正常现象,结块不会影响实验结果。如果不想结块,可以选择静置孵育(Rotate可能会增加结块的可能性,但不会影响结果),静置会看到沉降,也不影响结果。如果不放心,可以摇晃或者rotate。
一抗时间都可以,取决于时间安排。
4.pAG-Mnase酶结合:
问:4℃旋转1h,可延长吗?
答:1h已可使pAG与抗体进行结合,延长没有明显意义。
5.DNA消化和释放:
问:Spike-in DNA在NGS测序中是必须的吗?是qPCR中是必须的吗?
答:根据需求使用,spink in 不是必须的。
问:stop buffer室温化还是冰上化呢?
答:Stop buffer中有个物质,较为sensitive, 室温化,看到稍剩一点冰后,晃晃放冰上。
6.建库:(#56795 SimpleChIP® ChIP-seq DNA Library Prep Kit for Illumina/ E7645 NEBNext® Ultra™ II DNA Library Prep Kit for Illumina®)
建库前准备
问:AMpure beads和SPRI Select beads有区别吗?为什么AMpure beads至少需要提前在室温放置半小时呢?
答:没有区别,beads在常温下效率高。
问:End prep enzyme mix和End prep enzyme buffer是可以室温化吗?
答:前者不会冻起来,冰上,不可常温。buffer可以常温,vortex完全化开。
问:制备1XTE中1mM EDTA是必须的吗?作用是什么呢?
答:不是。起始DNA在TE中,故接着用TE。EDTA也可以换tris hcl。
接头蛋白连接
问:针对Cut&Run后无法定量的样本(低于Qubit检测范围),目前没有更微量仪器,如何稀释adaptor和选择PCR扩增循环数呢?有对应于细胞数的PCR循环推荐吗?比如我们现有5X10^4细胞,选用抗体为#9733 H3K27me3、#8173 H3K27ac、#9751 H3K4me3,可以选用怎样的稀释比和循环数呢?
答:产量靠经验,但也取决于Ab效率。
50K细胞,15-18 cycle ,根据Library DNA 浓度调整,library DNA浓度在10-30ng/uL都可以(如果没有picogreen,只能根据Library DNA 估算)。
需要Picogreen,用最多的20cycle, 或者先P12-14cycle,取2uL picogreen测浓度。不可用nanodrop,nanodrop需纯DNA。
问:Input的adaptor稀释问题:是根据实际的DNA浓度进行稀释还是选择和目的抗体样本管相同的adaptor稀释比呢?
答:根据实际浓度。
问:PCR仪的盖子设置为≥47℃,有具体的上限要求吗?是越高越好吗?
答:不是,盖子要高于酶切反应的37度,防止evaporation。47℃即可。
问:DNA end prep:推荐Cut&Run样本用到的20℃ 30min,50℃ 30min是适用于所有细胞量的吗?组蛋白、 转录因子都可以用这个条件吗?期间热盖温度应该设置多少呢?
答:跟细胞量无关,跟片段大小有关,适用于所有的cut run 片段。温度太高可使dna变性。根据protocol设置热盖。
片段清除
问:两步片段纯化中推荐的1.1X 纯化beads是适用于所有细胞量的吗?0.9X纯化beads你们有测试过吗?组蛋白、转录因子都可以用这个条件吗?
答:与DNA片段大小有关,buffer中APG的量决定了回收的是小片段还是大片段,浓度越高,回收的片段越小。CUT&RUN产物连adaptor 在200bp,左右 故1.1X OK。
问:开盖晾干beads的过程中如果晾干不够会对测序结果有影响吗?
答:乙醇影响PCR,太干损失DNA。Fang的操作:吸取上清后,开timer 5min,用小枪头再吸一遍(一般可以吸走2-3uL),等Timer到时间,加TE。
PCR扩增
问:对应于input样本可以选用相同的cycle数吗,还是根据所用的实际量比如5ng进行选择呢?
答:input根据实际量。见56795新版protocol。
问:PCR循环数设置中17cycle以上都需要进行分两步PCR吗?是因为Q5酶活性只能到达一定的cycle吗?两步中间需要定量吗?
答:最多20个循环,不做两次扩增了。
问:PCR扩增cycle数过多会有影响吗?
答:PCR会影响低丰度Peak不被读取(优先P高丰度),故Library DNA的量需控制。
问:如果按照上述分两次进行扩增,第一次扩增后也是选用1.1Xbeads进行纯化最后取15ul加到PCR体系吗,体系其它的量都是一样的是吗? 期间需要进行定量吗?
答:不做两次扩增了。
问:退火延伸时间是将建库kit里推荐的75s改为13s用于Cut&Run样本是吗?13s的退火延伸时间是适用于所有细胞数所有蛋白吗?
答:13s可以足以扩到1KB,希望小片段得以扩增,而非大片段。(10-15sec都可以)(此方法作为size selection的替代)
问:终末延伸65℃ 5min是适用于所有细胞数所有蛋白吗?
答:跟细胞数无关, 65℃ 3min也可。
问:该扩增条件对于input样本是和目的样本一致的吗?
答:是的。
PCR纯化
问:#56795里提到最终得到的DNA库的浓度10-40ng/ul是对应于多少细胞数呢?
答:5万-10万细胞所得浓度差不多,都应该到这个量。
问:具备多少量是可以送测序的呢?
答:10-40ng/uL, 应足够测序量(测序是nM级),测序公司希望收到浓样本,以便定量和由他们稀释。
问:推荐的测序深度是多少呢(我们用到的是双端测序)?
答:3-5M(protocol), 或者5-8即可。(M-million reads)如果希望最少的3M,则target 5Million,以平衡pippet和操作误差。
问:Protocol里写到的稀释所建DNA文库至5-10ng/ul是必须的吗,因为我们比较担心会和测序公司的质控浓度有偏差?
答:这是Bioanalyzer测试所需的浓度,5-10是较妥的。
测序质控及分析问题:
问:对于Bioanalyzer质控,组蛋白峰应该在什么位置是正确的呢?选用抗体为#9733 H3K27me3、#8173 H3K27ac、#9751 H3K4me3,对于转录因子峰应该在多大处呢?
答:300bp左右。转录因子200-300。
问:如果存在接头污染的问题也是可以上机的是吗,接头污染的peak和目的peak在Bioanalyzer质控中是可以区分的吗,接头污染的峰在什么位置呢?
答:接头污染可导致reads拿到的几乎都是接头。一点点污染可以接受,如结果中有5% adaptor也OK。可以区分。
问:Cut&Run的后续分析和chip-seq的后续分析是相似的吗?是否有推荐的pipeline呢?
答:IGV track等操作一样,分析可以尝试Henicoff的软件, 或其他。



