m6A甲基化修饰已成为近年来科研圈的“当红炸子鸡”,众所周知,m6A (N6-methyladenosine)是哺乳动物体内最为普遍的一种转录后RNA修饰,也是mRNA和非编码RNA中主要的甲基化修饰之一,它可以影响RNA的剪接、翻译、稳定性以及某些非编码RNA的表观遗传影响。
△点击放大图片
对于m6A甲基化的研究,通常是围绕着以下三种蛋白展开的(1)mRNA上存储m6A的甲基化转移酶复合物(Writers);(2)去除相关标志物脱甲基化酶(Erasers);(3)由m6A招募,且可对RNA发挥作用的甲基化结合蛋白(Readers),下表为大家整理了这三类甲基化酶常见的类型及其功能:

△点击放大图片
m6A甲基化涉及到很多不同的研究领域,尤其是肿瘤发生和癌症等研究领域具有众多发现,下表给大家整理了一些相关文献:
| 时间 | 期刊 | 相关疾病 | 相关m6A酶 | 参考文献* |
| 2018 | Nat Cell Biol. | 人子宫内膜癌 | METTL3 | -1 |
| METTL14 | Nat Cell Biol. | 人子宫内膜癌 | METTL3 | -1 |
| 2019 | Biochem Biophys Res Commun. | 人骨肉瘤 | METTL3 | -2 |
| 2019 | J Exp Clin Cancer Res. | 肾癌 | METTL14 | -3 |
| 2020 | Mol Cancer. | 胰腺癌 | ALKBH5 | -4 |
| 2020 | J Cell Mol Med. | 脑动静脉畸形 | WTAP | -5 |
| 2020 | Mol Cancer. | 结直肠癌 | METTL3 | -6 |
| 1GF2BPs | Mol Cancer. | 结直肠癌 | METTL3 | -6 |
| 2020 | Nucleic Acids Res. | 卵巢癌 | EIF3C | -7 |
| 2021 | Nat Cancer. | 乳腺癌 | FTO | -8 |
| WNT | Nat Cancer. | 乳腺癌 | FTO | -8 |
| 2022 | Nat Commun. | 结直肠癌 | METTL3 | -9 |
| 2022 | J Gene Med. | 肺癌 | METTL3 | -10 |
| ALKBH5 | J Gene Med. | 肺癌 | METTL3 | -10 |
| YTHDC2 | J Gene Med. | 肺癌 | METTL3 | -10 |
| YTHDF1 | J Gene Med. | 肺癌 | METTL3 | -10 |
* 具体文献请见文末
接下来小优为大家选取了具有代表性的两篇高分文献(分别发表在《Molecular Cancer》和《Nature Communication》期刊上),从实验方法和主要研究结果两个维度进行分析,希望能给大家的科研思路带去一些参考:
PART1 RNA去甲基化酶ALKBH5以m6a - YTHDF2依赖的方式通过PERT的转录后激活来阻止胰腺癌的进展

△点击放大图片
研究背景
胰腺癌(pancreatic cancer,PC)是一种发病隐匿、诊断困难、手术切除率低的高度恶性胃肠肿瘤,已成为世界范围内最常见的致死性疾病之一,其5年生存率仅为8%。因此,迫切需要阐明PC背后的分子和细胞机制,以达到诊断和治疗干预的目的。m6A是真核生物mRNA中含量最多的可逆甲基化修饰,早被证实其参与肿瘤的发生发展,但关于其对于胰腺癌的作用机制尚不明确。作者在这篇文章中重点探讨了m6A去甲基化酶ALKBH5在胰腺癌中的作用。
实验方法
实验方法主要包括以下5个部分:
❖从PC病人中收集了42个PC样本,并使用real-time PCR、WB和IHC实验对其ALKBH5的表达及临床病理影响进行评估;
❖在获得功能和失去功能分析的基础上,使用RNA测序研究ALKBH5对PC细胞生物学特性的影响;
❖使用流式细胞术、WB等对小鼠样本的皮下和原位模型进一步研究,用于揭示ALKBH5在肿瘤生长中的作用;
❖通过MeRIP-qPCR(RNA甲基化测序-qPCR)对mRNA和m6A进行测序用于确定ALKBH5对PER1的靶向作用;
❖通过ChIP和荧光素酶检测ALKBH5启动子中的p53结合位点,揭示ALKBH5与PER1激活的ATM-CHK2-P53/CDC25C信号通路之间的相互作用。
主要实验方法及结果
01 ALKBH5的损失是PC的特征,具有预测疾病和预后的价值
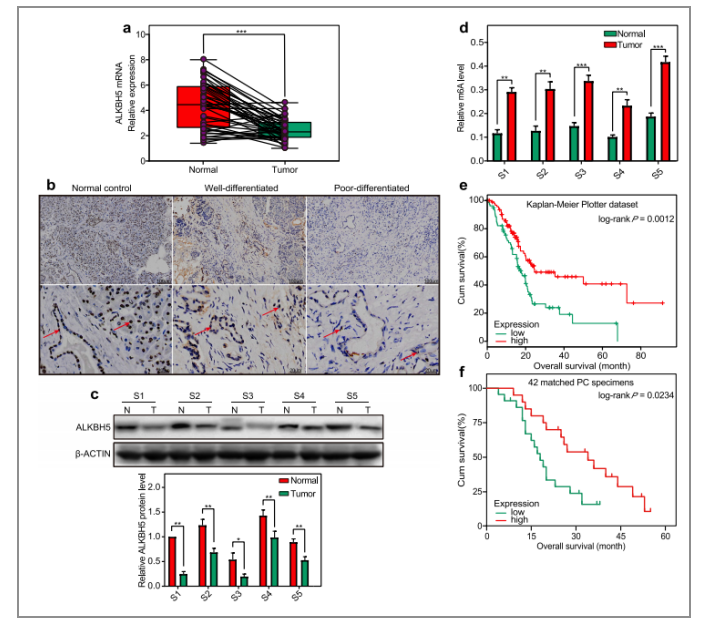
图1. m6A去甲基化酶ALKBH5的下调是胰腺癌的特征
△点击放大图片
作者首先分析了42个PC样本的临床表现(IHC-图1/b,WB-图1/c,RT-qPCR-图1/d),结果发现ALKBH5在PC患者的样本中表达量均低,且这些低表达量的样本中m6A的水平较高。图1/e使用Kaplan-Meier Plotter进行了生存曲线分析,结果显示ALKBH5低表达的患者预后较差。
02 ALKBH5可抑制PC细胞增殖、迁移和侵袭,并且可以让细胞滞留在G2/M期(体外实验)
为了确定ALKBH5依赖的m6A去甲基化的作用,作者分别对敲除/过表达ALKBH5基因的细胞进行了多角度的分析:
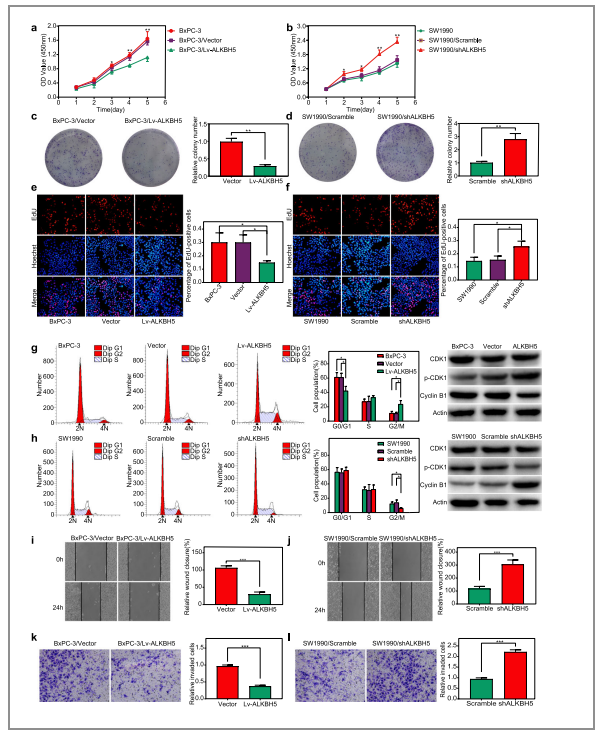
图2. ALKBH5对PC细胞的增殖、细胞周期、迁移和侵袭的抑制作用
△点击放大图片
(1)使用商业化的细胞增值检测盒(CCK-8、EdU)来评估PC细胞的增殖活性(图2/a,e),结果显示ALKBH5的过表达可以显著地减少PC细胞的增殖;利用菌落形成试验(Colony formation assays)也可以观察到ALKBH5的过表达削弱了PC细胞菌落的长期形成,ALKBH5的敲除则呈现了相反的结果;
(2)作者将流式细胞术和WB实验进行结合(图2/g,h),观察到ALKBH5的过表达(Lv-ALKBH5组)导致BxPC-3细胞在G2/M期的百分比显著增加,而ALKBH5的下调(shakbh5组)使得G2/M期细胞数量明显减少;
(3)在单层PC细胞上进行伤口愈合试验和transwell实验分别来评估PC细胞的迁移和侵袭能力(图2/i,j,k,i),结果显示ALKBH5的过表达会抑制PC细胞的这两种能力。
03 ALKBH5可抑制PC的生长和转移(体内实验)

图3. ALKBH5抑制PC模型的肿瘤生长和侵袭潜能
△点击放大图片
作者接着构建了ALKBH5敲减/过表达的异种移植瘤小鼠模型和原位瘤小鼠模型,通过形态学、IHC和生物发光呈象(BLI)等角度进行了分析。
(1)与低表达ALKBH5的PC组织相比,过表达的肿瘤组织的质量有着明显的下降(图/a,c,d,f);
(2)免疫组化实验结果显示,ALFBH5过表达会造成ki-67(体内实验中细胞生长的关键指标之一),MMP-2 和 MMP-9的下调,而ALFBH5的敲除则会造成反效果(图/g,h,i,j);
(3)使用BLI技术在原位瘤小鼠模型中研究发现,植入BxPC-3/Lv-ALKBH5细胞的裸鼠荧光素酶活性明显低于植入BxPC-3/载体细胞的裸鼠;携带SW1990/shALKBH5细胞的小鼠荧光素酶信号明显高于携带SW1990/scramble细胞的小鼠(图/k,l)
04 ALKBH5的靶基因是PER1,且ALKBH5以m6A - YTHDF2依赖的方式增加PER1 mRNA的水平
为了进一步确认ALKBH5对PC细胞的转录谱是怎么影响的,作者对总共367个差异表达的基因(有204个上调的基因,163个下调的基因)进行了RNA-seq分析。GO和KEGG的结果均表明了,在ALKBH5过表达的PC细胞中,那些会造成细胞凋亡的基因均为上调基因,而那些造成细胞生存和增值的基因为下调基因(图4)。这些结果表明了ALKBH5可能通过影响基因转录参与调节PC。
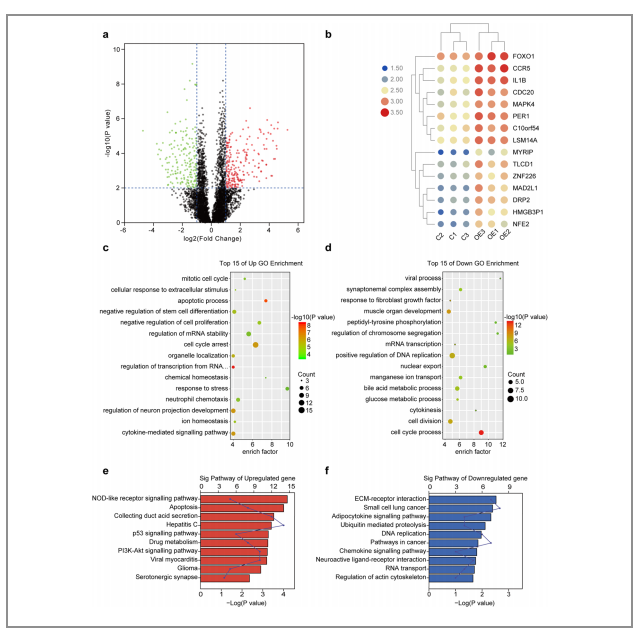
图4. ALKBH5对PC细胞转录谱的影响
△点击放大图片
作者在整体分子水平上对ALKBH5进行了分子调控可能性的分析之后,又进行了m6A-seq的测序,以确定ALKBH5的潜在靶基因。作者总共筛选出了9个高甲基化的靶基因(图5/e),其中发现PER1 mRNA的3 ' -UTR中有一个m6A峰值的富集,该峰值在ALKBH5的过表达降低后减少(图5/f),这暗示了ALKBH5是可能的靶基因。
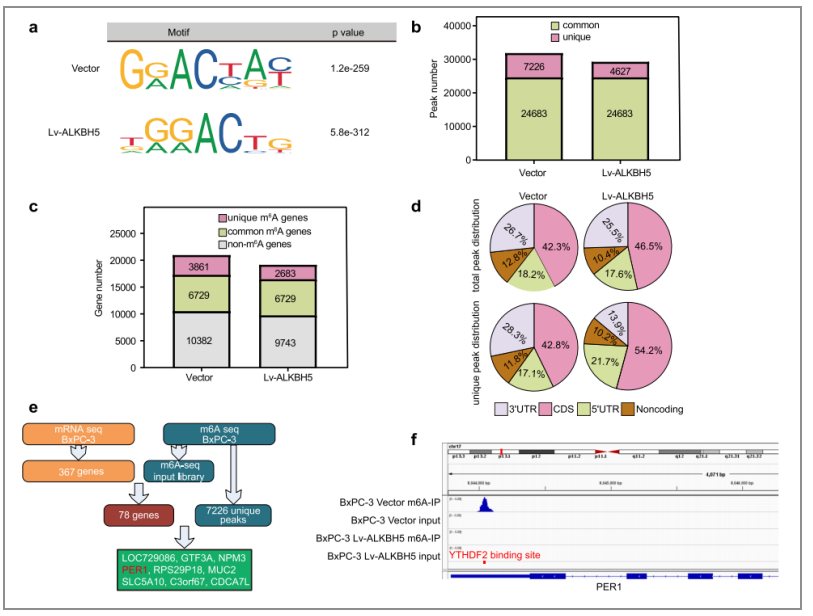
图5. m6A甲基化是ALKBH5起作用的基础
△点击放大图片
为了进一步确认PER1是ALKBH5的靶基因,作者对ALKBH5基因进行了敲除和过表达,结果显示敲除后的PC细胞显示低的PER1甲基化水平,而过表达后的PC细胞则显示高的PER1甲基化水平(图6/a)。通过MeRIP-qPCR实验,证明了ALKBH5可调控PER1的mRNA水平及m6A甲基化水平(图6/b)。对PER1进行序列分析后,PER1 mRNA中的m6A修饰位点位于YTHDF2结合位点附近(图5/f);在敲低YTHDF2后,PC细胞中PER1 mRNA的水平上升(图6/e)。因此,ALKBH5被认为通过消除m6A- YTHDF2依赖的mRNA降解来增加PER1 mRNA水平。

图6. ALKBH5以m6A - YTHDF2依赖的方式增加PER1 mRNA水平
△点击放大图片
05 靶基因PER1对PC有抑制作用
在确定了ALKBH5的靶基因是PER1之后,作者又对PER1的生物学功能进行了分析。
(1)42个临床样本分析发现,PC患者组织中PER1基因有所上调;
(2)通过免疫荧光实验发现,可发现PER1的过表达可以减少因ALKBH5的敲除而引起的细胞形态上的变化,证明了PER1和ALKBH5的表达为正相关(图7/e);
(3)细胞增值检测盒(CCK-8、EdU)和transwell检测证实,PER1的异位表达抑制了PC的进展,并部分挽救了ALKBH5基因下调后PC细胞增殖和侵袭过程中观察到的异常状态(图7/f,g,h);
(4)WB实验结果显示,在PER1缺失的前提下(敲除ALKBH5引起的),G2/M期细胞周期阻滞基因的下调和CYCLIN B1的上调,而PER1的过表达部分逆转了这些异常,证实了正常表达的PRE1可以重新激活ATM的相关信号通路(图7/i);
(5)CoIP实验则进一步证实了PER1和ATM之间的关系(图7/j)

图7. ALKBH5与PC组织中PER1表达相关,具有抑癌的作用
△点击放大图片
06 PER1通过诱导P53从而恢复ALKBH5转录的上调
(1)P53(与ATM相关的基因之一)的异位表达使得PC细胞中ALKBH5的水平有了显著地提高(图8/a,b);
(2)同时免疫荧光实验结果也显示了P53的表达降低了PC细胞中RNA的甲基化水平(图8/c);
(3)接着作者设计了两段引物用于覆盖P53两段的结合位点,并在转录了P53基因的PC细胞中进行ChIP实验,发现P53能与两个位点结合,其中绝大多数结合在位点1(图8/e);
(4)进一步进行荧光素酶的检测发现P53通过野生型启动子刺激ALKBH5的表达,而这种效应则被位点1或位点2的突变所抵消(图8/f,g)。
以上实验均证实了PER1可以通过P53基因来控制ALKBH5的转录
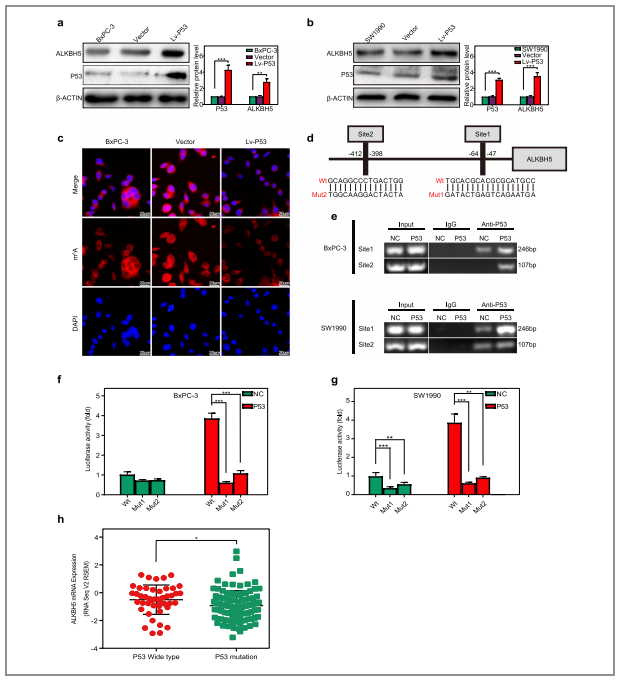
图8. per1通过诱导P53反馈调节ALKBH5的转录
△点击放大图片
结论
ALKBH5作为PC的抑制因子,可通过m6A的消亡来调节PER1的转录后激活,这为后续的基于去甲基化来进行PC诊断和治疗的研究提供了更多可能性。

图9. ALKBH5诱导PER1转录后激活,以m6A- YTHDF2依赖的方式抑制PC进展
part2 形核梭杆菌 (Fusobacterium nucleatum)可以減少METTL3介导的m6A修饰井促进结直肠癌转移

△点击放大图片
研究背景
微生物和宿主相互作用在结直肠癌(colorectal cancer,CRC)的进展中起着至关重要的作用,许多前期的实验证明细菌的病原体可以通过多种方式调节宿主的表观基因组学,其中RNA m6A甲基化是至关重要的一种。m6A修饰及其相关蛋白的失调已显示出对人类癌变的显著影响,但微生物-宿主互作用是否影响宿主mRNA的m6A修饰及其潜在机制仍不明确。
主要实验方法及结果
01 形核梭杆菌在人CRC细胞和PDX组织中可以减少METTL3介导的m6A修饰
作者使用HCT116细胞建立了CRC细胞模型并从CRC病人中获取了人源肿瘤异体移植模型(PDX)模型,并进行核梭杆菌处理。
(1)通过Dot blot、和UHPLC Q-Exactive质谱分析观察到CRC细胞与形核梭杆菌共培养后,CRC细胞里的m6A和mRNA m6A的修饰水平都显著地降低了(图10/a,b,c);
(2)通过WB实验可以发现,经过形核梭杆菌处理过的CRC细胞显著降低了METTL3的表达水平,但是并没有观察到对于其他m6A erasers或readers的影响(图10/e);
(3)在PDX模型里也观察到了一致的结论:METTL3 mRNA在经受形核梭杆菌处理过的PDX中水平显著降低(图10/g,h)。
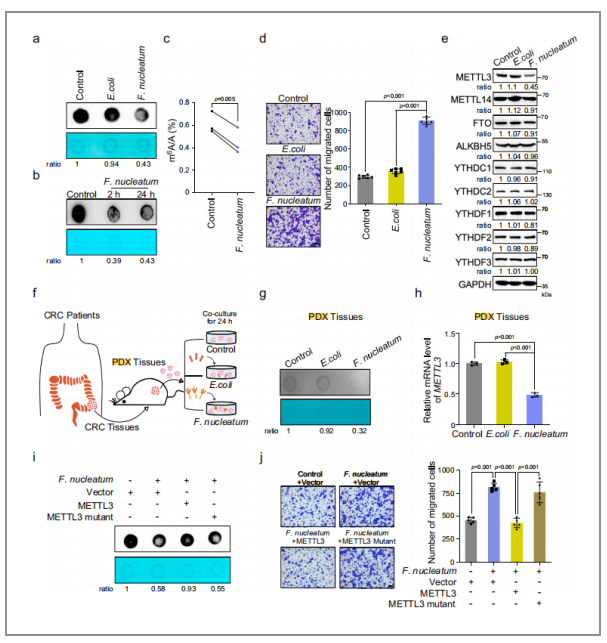
图10. 形核梭杆菌在人CRC细胞和PDX组织中减少METTL3介导的m6A修饰
△点击放大图片
02 形核梭杆菌通过调节Hippo-YAP信号通路来抑制METTL3
为了探索形核梭杆菌如何抑制METTL3并进一步调节m6A修饰的稳态的机制,作者筛选了经形核梭杆菌处理后的CRC细胞中受到激活的信号通路。
(1)通过qRT-PCR可以筛选出CRC细胞中经形核梭杆菌处理后被激活的信号通路,实验结果显示与NF-κB和Hippo-YAP两个信号通路相关的基因在CRC细胞中均被激活了(图11/a,b);
(2)通过免疫荧光实验和荧光素酶的实验结果也进一步证实了经形核梭杆菌处理的CRC细胞中YAP的水平也显著的升高(图11/d,e);
(3)作者又通过WB实验证实了在经形核梭杆菌处理后的CRC细胞中,MST1/2 和 LATS1/2的磷酸化水平显著的降低(图11/f);
(4)通过Transwell实验想要进一步证实YAP信号通路是否可以通过调节形核梭杆菌的功能来促进CRC的聚集,实验结果显示YAP的敲除可以显著降低CRC细胞迁移的能力(图11/g)
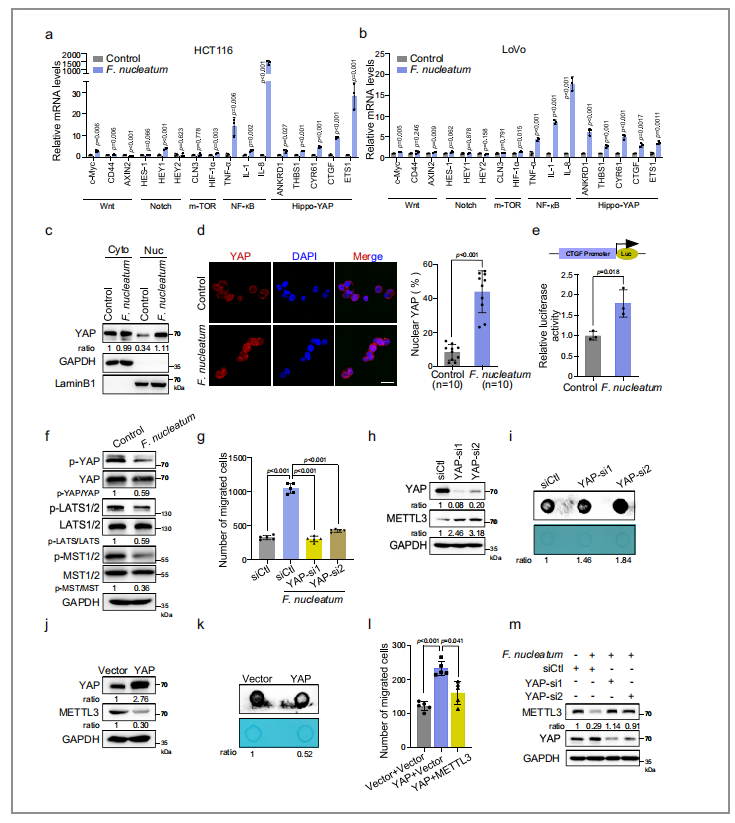
图11. 形核梭杆菌通过调节Hippo - YAP信号通路来抑制METTL3
△点击放大图片
03 FOXD3是METTL3的转录因子之一
接着作者想要探究YAP信号通路是通过什么机制来调节METTL3的表达,作者一开始想到的是通过TEAD转录因子(因为有研究证明YAP通过与TEAD转录因子的关联来调节基因转录),但是作者在METTL3启动子中未找到TEADs的结合位点。因此作者通过线上生物信息平台来预测了可能的转录因子,找到了FOXD3(图12/a)。
(1)通过WB实验观察到FOXD3的异位表达显著增加了METTL3的mRNA(结果中没有展示)和蛋白水平(图12/b);相反地,FOXD3敲除后的,METTL3的mRNA(结果中没有展示)和蛋白水平(图12/c)得到了显著降低;
(2)接着为了进一步证实FOXD3是METTL3的直接转录因子,作者进行了ChIP实验,结果表明了FOXD3与METTL3的启动子结合;在FOXD3-P1和FOXD3-P2这两个区域都可能包含假定的FOXD3结合元素(图12/f);
(3)双荧光素酶实验结果也证明了FOXD3的过度表达可以显著提高CRC细胞中METTL-荧光素的活性(图12/f)
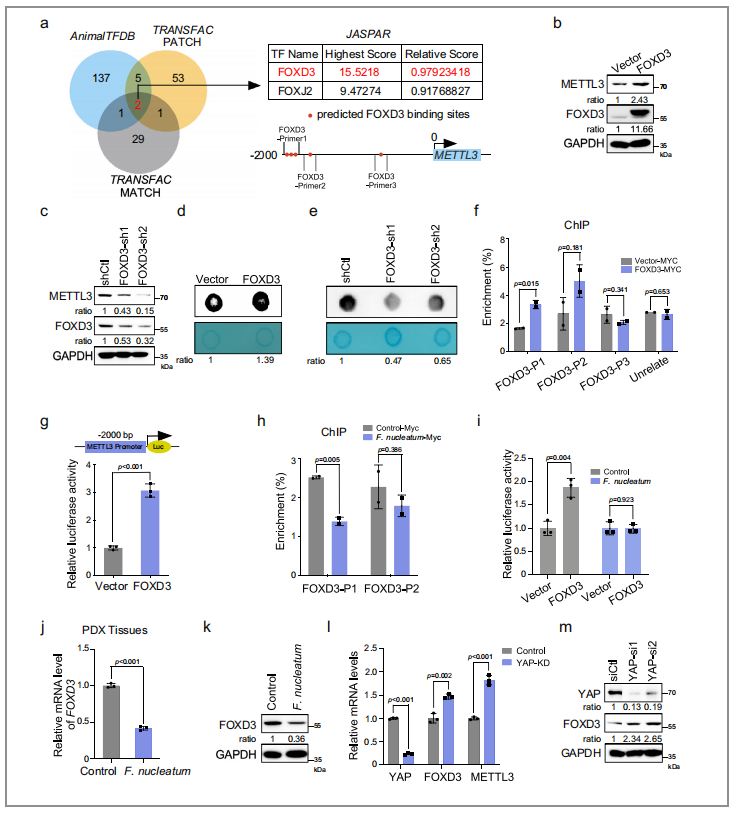
图12. FOXD3是METTL3的转录因子
△点击放大图片
04 通过m6A-seq和RNA-seq测序发现KIF26B是METTL3下游调控基因
为了研究m6A修饰在特定基因中的变化,作者在形核梭杆菌处理过的CRC细胞中进行了m6A-seq测序。发现有5841个m6A峰值在这些CRC细胞中发生了变化,其中有2835个上调的峰值,3006个下调的峰值。与上篇文章相反地,本文是在m6A-seq结果的基础上,再进行了整体水平上的mRNA分析。作者观察到在经形核梭杆菌处理后的细胞中,m6A在5'UTR附近出现峰值,而mRNA的CDS区域减少,尽管3'UTR区域保持相似的丰度。
KEFF结果显示了形核梭杆菌可通过多方面影响CRC细胞,如粘着性和Hippo信号通路(图13/d);GO结果则显示了这些细胞中,m6A修饰发生在少数与调节GTPase活性、细胞骨架组织和细胞极性重塑相关的基因上(图13/e)
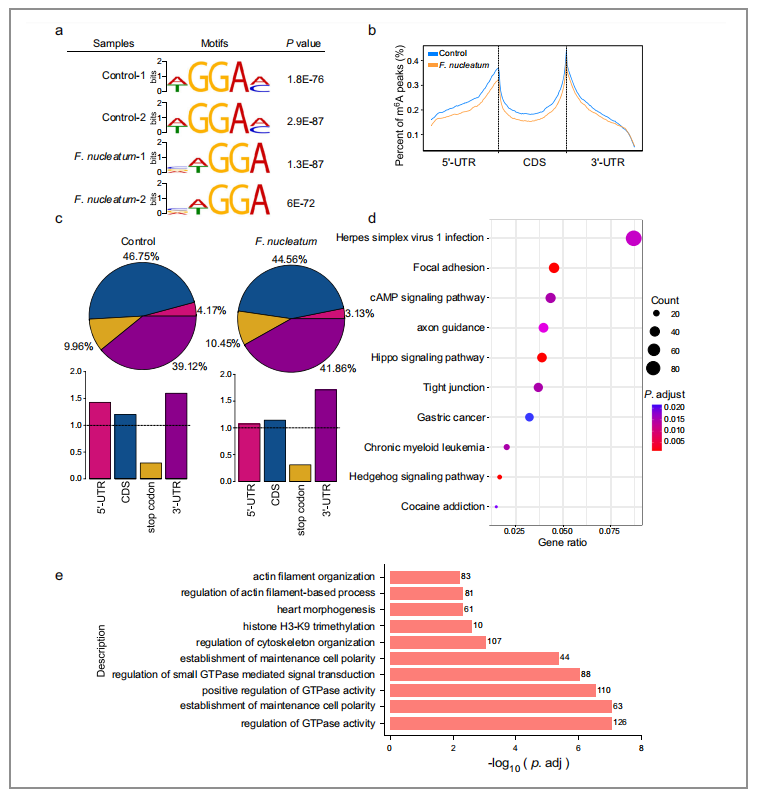
图13. 经形核梭杆菌处理后的CRC细胞中,m6A调控基因的变化
△点击放大图片
通过对比经形核梭杆菌处理的CRC细胞和对照组的热力图,发现155个上调的基因和88个下调基因,其中有5个基因是在m6A测序和mRNA测序中相重合后的结果(图14/a,b),在这5个基因中,作者初步是被出了KIF26B,因为其参与了细胞骨架识别(这个生物活动对于CRC的聚集十分重要)。
为了进一步证实KIF26B mRNA是METTL3的靶标,作者进行了RNA免疫沉淀实验(RIP),其中METTL3靶基因CREBBP做为阳性对照,而HPRT1做为阴性对照。结果显示KIF26B的表达受YAP/FOXD3/METTL3轴的调控,且KIF26B受核梭杆菌影响的上调也取决于METTL3的m6A甲基转移酶活动(图14/h,i,j)。

图14. 通过m6A-seq和RNA-seq测序发现,KIF26B是METTL3的下游靶点
△点击放大图片
以上实验结果均表明了形核梭杆菌诱导的METTL3下调降低了KIF26B的m6A修饰水平,并进一步降低了YTHDF2依赖的mRNA降解,从而促进了KIF26B在CRC细胞中的表达,因此证明了KIF26B是METTL3下游调控基因。
05 形核梭杆菌通过上调KIF26B来加速CRC的聚集和转移
据证实,KIF26B作为癌基因会参与胃癌和乳腺癌,但其在CRC中可能的调控机制还未被明晰,因此作者在CRC细胞中对KIF26B进行了敲除并进行了mRNA的测序(图15/a),初步证实了KIF26B与CRC的聚集有关联(图15/b)。
(1)通过m6A RIP-seq检测发现,在KIF26B基因的调控下,Hippo信号通路被显著地激活了(图15/c)
(2)同时,作者在经METTL3敲除后的CRC细胞中进行了KIF26B基因的沉默,接着通过WB实验发现(图15/d,f),由METTL3敲除而引起的KIF26B基因的缺失会减弱CRC细胞的迁移
(3)通过RIP-qPCR实验发现(图15/n)
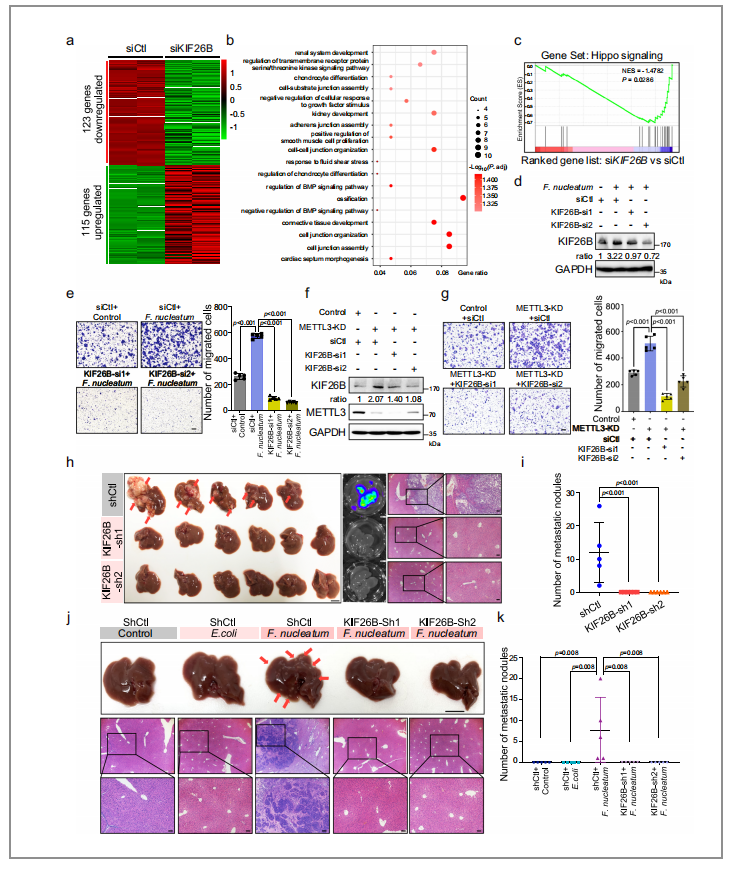
图15. 核梭杆菌通过上调KIF26B加速CRC的侵袭性和转移性
△点击放大图片
结论
经证实,核梭杆菌与CRC患者METTL3和KIF26B表达相关,具体的调节机制如图16所示:
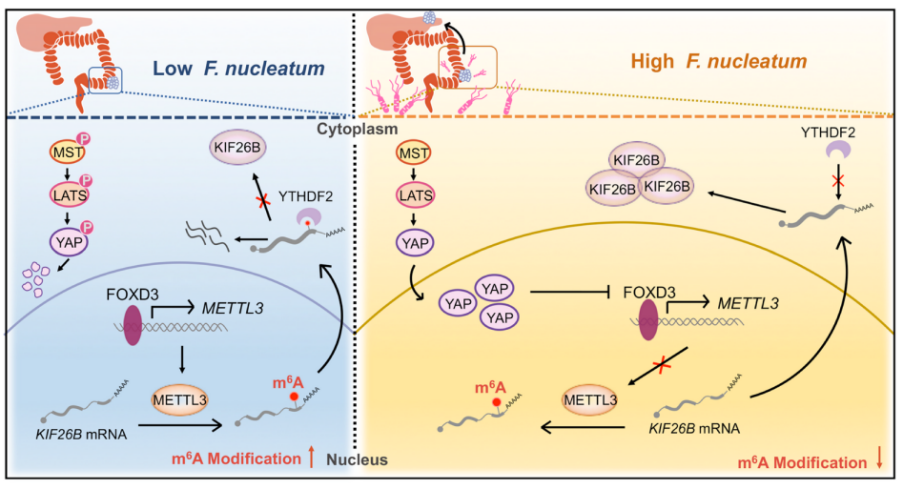
图16. 核梭杆菌诱导的METTL3下调m6A修饰有助于CRC的转移
△点击放大图片
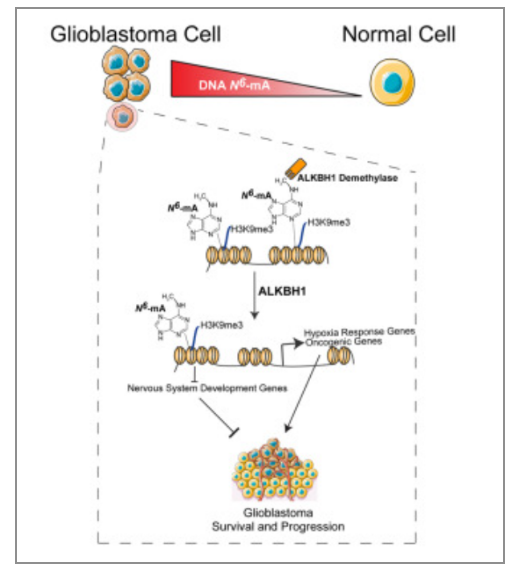
除了RNA甲基化的研究,m6A也被发现在DNA修饰中起到作用,早在2018年的《cell》期刊中,就发现了m6A的DNA修饰水平可以影响胶质瘤(doi:https://doi.org/10.1016/j.cell.2018.10.006),感兴趣的老师同学可以点击这里查看研究。
△点击放大图片
△点击放大图片
上海优宁维作为生命科学一站式服务商可以给广大科研工作者提供与m6A甲基化研究相关的试剂和软件:
| 类别 | 常见基因/蛋白 | 货号 | 应用 | 来源种属 |
| m6A | m6A(文献应用量达468篇优宁维常备现货!) | 202003 | Dot Blot, IP, ELISA | 兔 |
| m6A Writer | CBLL1 | sc-517157 | WB, IP, IF 和 ELISA | 鼠 |
| m6A Writer | METTL3 | sc-518180 | IP, WB, IHC(P),ELISA,IF,FCM | 鼠 |
| m6A Writer | METTL5 | HPA038223 | IHC,ICC-IF | 兔 |
| m6A Writer | METTL14 | HPA038002 | IHC,ICC-IF | 兔 |
| m6A Writer | METTL16 | HPA059798 | ICC-IF | 兔 |
| m6A Writer | RBM15 | HPA049642 | WB,ICC-IF | 兔 |
| m6A Writer | RBM15B | HPA036645 | IHC,WB,ICC-IF | 兔 |
| m6A Writer | VIRMA/KIAA1429 | HPA031530 | IHC,ICC-IF | 兔 |
| m6A Writer | WTAP | sc-166931 | WB, IP, IF, ELISA | 鼠 |
| m6A Eraser | ALKBH3 | sc-376520 | WB,IP,IF,ELISA | 鼠 |
| m6A Eraser | ALKBH5 | 80283 | WB | 兔 |
| m6A Eraser | FTO | HPA041086 | IHC,WB | 兔 |
| m6AReader | EIF3A | sc-376651 | WB,IP,IF,ELISA | 鼠 |
| m6AReader | EIF3B | sc-374156 | WB.IP,IF,IHC | 鼠 |
| m6AReader | EIF3C | HPA049495 | IHC,WB,ICC-IF | 兔 |
| m6AReader | EIF3D | HPA066216 | IHC,WB,ICC-IF | 兔 |
| m6AReader | eIF3γ | sc-271283 | WB.IP,IF | 鼠 |
| m6AReader | FMR1 | sc-101048 | WB,IP,ELISA | 鼠 |
| m6AReader | HNRNPA2B1 | HPA065537 | WB,ICC-IF | 兔 |
| m6AReader | HNRNPC | HPA051075 | IHC,WB,ICC-IF | 兔 |
| m6AReader | HuR | sc-5261 | WB, IP, IF, IHC(P), ELISA | 鼠 |
| m6AReader | LRPPRC | HPA036409 | IHC,WB,ICC-IF | 兔 |
| m6AReader | RBMX2 | HPA026609 | IHC,WB,ICC-IF | 兔 |
| m6AReader | YTHDC1 | HPA036462 | IHC,ICC-IF | 兔 |
| m6AReader | YTHDC2 | HPA072678 | ICC-IF | 兔 |
其他部分相关产品
| 货号 | 产品 | 规格 | 分类 |
| sc-377293 | Atm (G-12) | 200ug/ml | 一抗 |
| sc-23900 | Ki67 (Ki-67) | 200ug/ml | 一抗 |
| sc-398890 | Per1 (E-8) | 200ug/ml | 一抗 |
| sc-71811 | p21 Waf1/Cip1 (0.N.488) | 200ug/ml | 一抗 |
| 111-005-003 | AffiniPure Goat Anti-Rabbit IgG (H+L) | 2mg | 二抗 |
| 51304 | QIAamp DNA Mini Kit | 50test/250test | 试剂盒 |
| 218073 | miScript SYBR Green PCR Kit | 1000test/2000test | 试剂盒 |
| 28106 | QIAquick PCR Purification Kit | 50test/250test | 试剂盒 |
| abs50003 | CCK-8试剂盒 | 1ml/5ml/50ml | 试剂盒 |
| SH30023 | DMEM | 500ml/1000ml | 培养基 |
| LS002004 | Deoxyribonuclease I | 5mg/20mg/100mg/bulk | 酶 |
| 13884 | Puromycin | 25mg/50mg/bulk | 小分子化合物 |
| sc-2003 | Protein A/G PLUS-Agarose | 2ml | 辅助试剂 |
| sc-3877 | normal mouse IgG1 | 200ug/ml | 辅助试剂 |
本文中的两篇文献都是采用Graphpad Prism 9.0绘制的,这是一款数据分析和可视化软件,为科学研究而设计。它集生物统计,曲线拟合和科技图于一体有超过10国家的科学家们依靠Graphpad 软件来分析,绘制和展示他的科学数据。该软件可提供1年期、永久版、单用户、多用户等多种版本,详情请点击此处了解。
参考文献:
(1) m6A mRNA methylation regulates AKT activity to promote the proliferation and tumorigenicity of endometrial cancer
(2) The m6A methyltransferase METTL3 promotes osteosarcoma progression by regulating the m6A level of LEF1. Biochem Biophys Res Commun.
(3) The m6A-suppressed P2RX6 activation promotes renal cancer cells migration and invasion through ATP-induced Ca2+ influx modulating ERK1/2 phosphorylation and MMP9 signaling pathway.
(4) RNA demethylase ALKBH5 prevents pancreatic cancer progression by posttranscriptional activation of PER1 in an m6A-YTHDF2-dependent manner
(5) Wilms' tumour 1-associating protein inhibits endothelial cell angiogenesis by m6A-dependent epigenetic silencing of desmoplakin in brain arteriovenous malformation.
(6) m6A-dependent glycolysis enhances colorectal cancer progression.
(7) The m6A reader YTHDF1 promotes ovarian cancer progression via augmenting EIF3C translation.
(8) Downregulation of the FTO m6A RNA demethylase promotes EMT-mediated progression of epithelial tumors and sensitivity to Wnt inhibitors.
(9) Fusobacterium nucleatum reduces METTL3-mediated m6A modification and contributes to colorectal cancer metastasis
(10) The pathological tissue expression pattern and clinical significance of m6A-regulatory genes in non-small cell lung cancer.