△点击放大图片
白
呼叫罗工,最近在看文献,发现文章中有大量的这种流式导图或者脑图,为什么不用散点图呢?
罗
流式数据分析我们习惯用传统的直方图或者二维散点图进行数据的展示,现在越来越多人开始使用降维或者聚类的算法分析,比如t-SNE降维分析。
案例:B细胞分群(SLE疾病相关,检测指标包含 IgD, IgM,CD5, CD10, CD19, CD21, CD24, CD27和 CD38),该实验作者分为两个批次,各取16和20个人(包括疾病和正常人)的外周血样本提取PBMC,如果每个样本都要如此分析,需要大量的时间和重复工作。
散点图结果
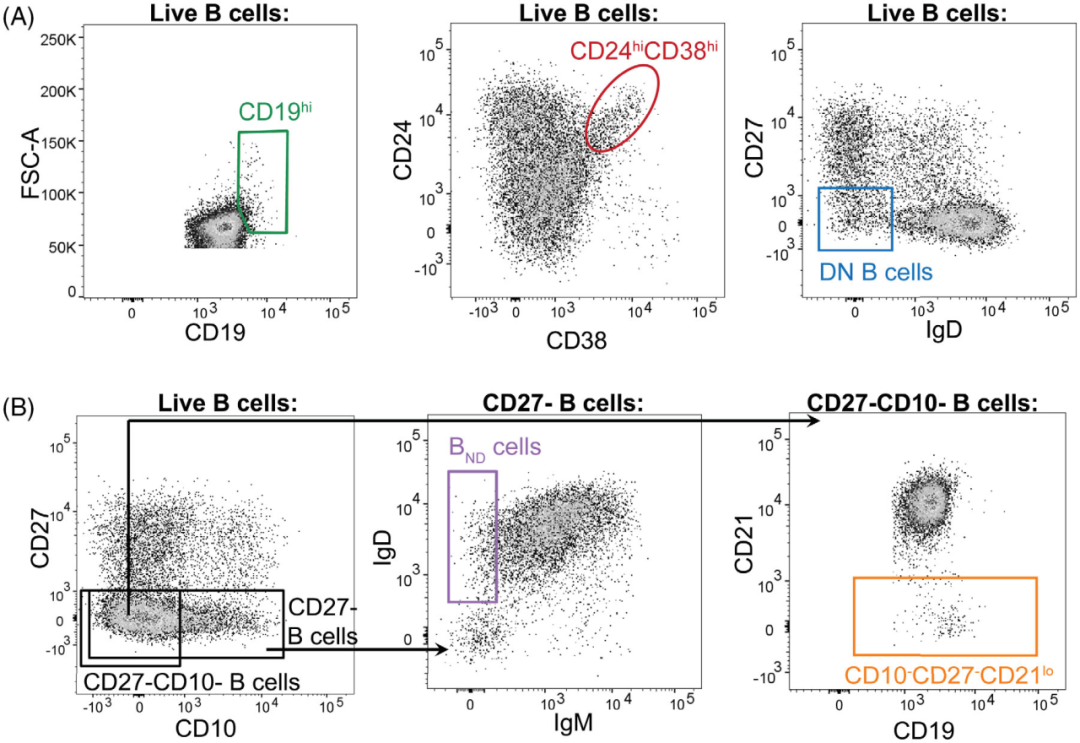
△点击放大图片
但是使用T-SNE降维分析等大数据分析能够将自身免疫相关B亚群的异质性、能够将不同的群体非常Easy的展示出来。
自身免疫相关B亚群的异质性

△点击放大图片
B与自身免疫易感亚群重叠
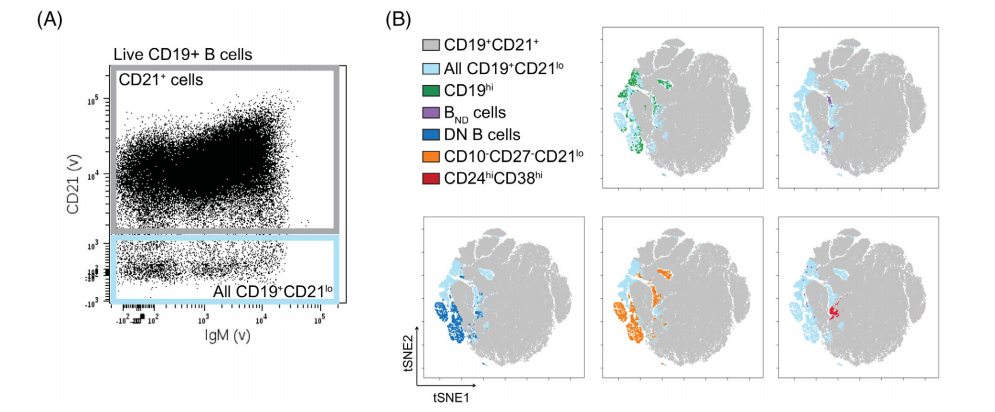
△点击放大图片
CD19+CD21lo B细胞的异质性;
换成HeatMap&旭日图表达输出

△点击放大图片
白
但是罗工,降维聚类插件那么多,我该如何选?
罗
其实降维聚类没有说一定要选哪种方式,哪种组合最好,是根据自己的数据情况,降维聚类后,我们主观认为哪种组合最好。
但总的来说tSNE和UMAP文献使用率最高,tSNE图比较饱满,对亚类分群更突出,比方Naive、Memory Treg都能区分,UMAP对分化展示 维只是生成一个平面,呈现所有检测的细胞的聚类分布。
但我们要提取出每群聚类在一起的数据,需要聚类插件帮助,首推X-shift,它聚类群体数适中,Phenograph提取的群体数太多,FlowSOM需要指定群体数,可以在X-shift分群基础上设置FlowSOM的分群数和分群参数进行优化分群。
当然我们也可以把我们手动设门策略,在降维图上进行Overlap。
降维:tSNE、UMAP、TriMap、PaCMAP
聚类:FlowSOM、Phenograph、XShift
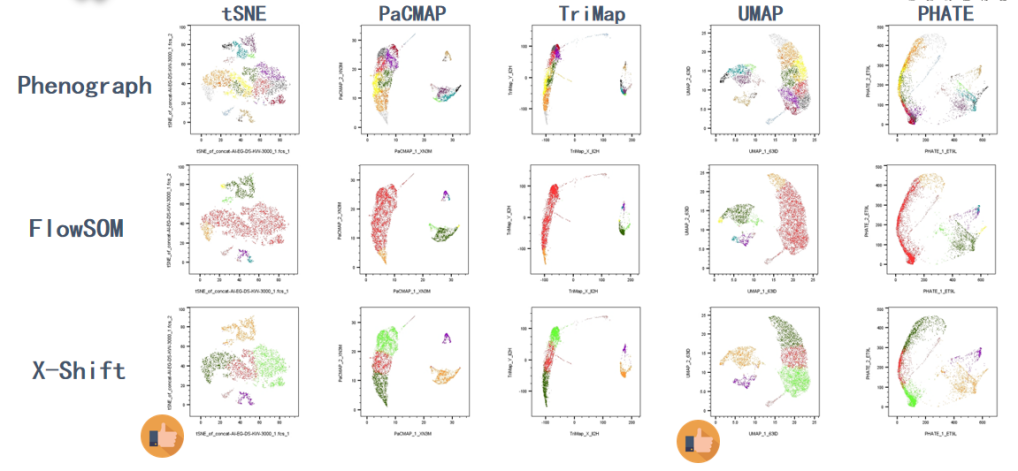
△点击放大图片
问
罗工,每一群在这个降维图上分出来,怎么知道他们是什么细胞呀?
答
降维聚类,是帮助我们展示流式数据,但群体注释,是什么指标差异导致分群,还需我们自己比对注释。
但也不必担心工作量的问题,FlowJo有ClusterExplorer和MEM帮助我们。
ClusterExplorer包含表达矢量图、降维图、热图、群体占比图、和设置。
自动聚类将样本分成多少群,在表达矢量图上的每个参数的矢量尺上就有多少个点,这个点就是自动聚类的细胞群的该参数的平均值,我们根据自己感兴趣的目的细胞群,检测蛋白指标在样本中的表达量进行圈选,就可以找到对应的Cluster,对应的降维图、热图、群体占比图都会凸显出来。
探索对比:ClusterExplorer
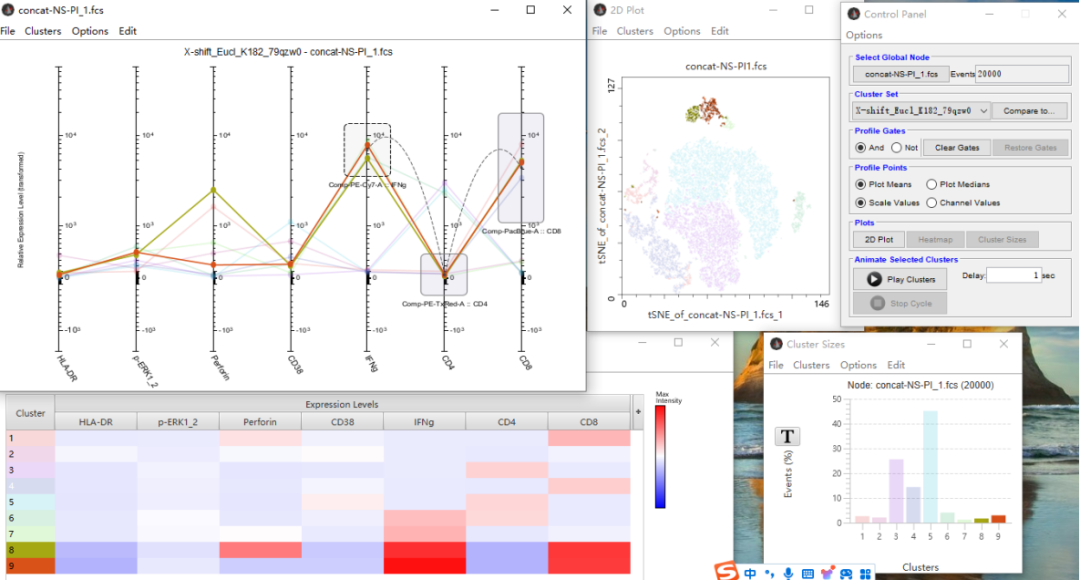
△点击放大图片
MEM是在主页面里,对自动聚类生成的数据条进行自动注释的工具,直观告诉我们每个聚类出的Cluster显著差异的检测参数表达相对量,+表示阳性,数字表示相对表达量,数值越大,表达量越高。
注释合并:MEM、人为定义

△点击放大图片
白
FlowJo真的是太强大了,FlowJo发现新细胞群后,想分选出来做一些功能实验能实现吗?
罗
当然,这是荧光流式在其他高维技术中的优势所在,可以分选到活细胞。
白
那这个降维图咋分选,而且这个图是我前面实验的,我要分选又是新样本数据,还有分选软件也对应不起来。
罗
不必担心,Embed这个插件可以将新数据投射到之前的降维聚类分析里,快速找到目标细胞群。软件不匹配,可以用HyperFinder,快速生成分选设门策略,通过FlowJo对接Diva分选平台,其他非Diva软件的分选仪得自己手动圈门HyperFinder生成的分选设门策略。
新数据投射:Embed
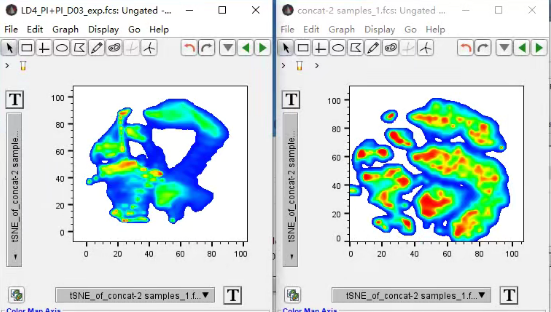
△点击放大图片
分选圈门:HyperFinder
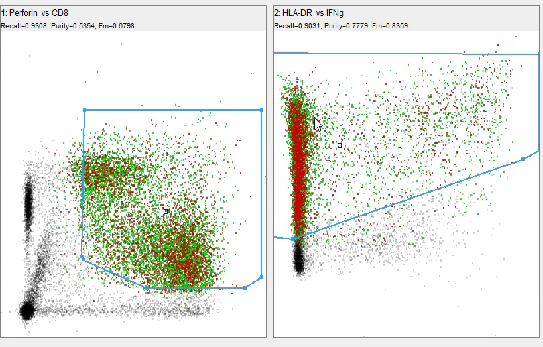
△点击放大图片
最后给你一个总结高维流式分析的操作流程,按照这个流程保管分析不出错!
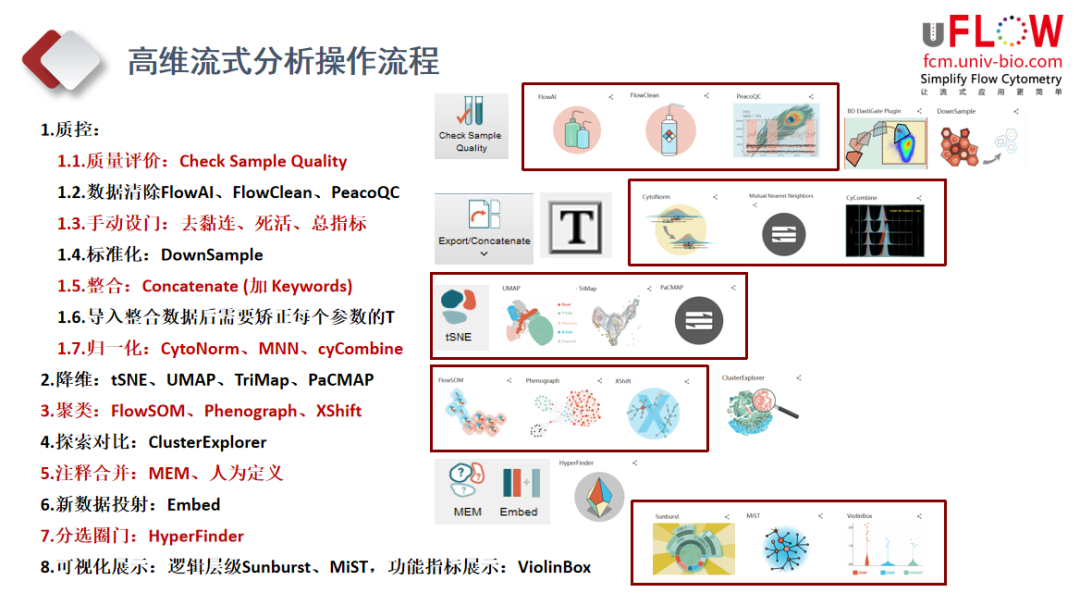
△点击放大图片