染色质免疫沉淀(ChIP)一直是研究蛋白质-DNA相互作用的主要方法,主要包括细胞与甲醛交联固定、染色质片段化、引入抗体富集、收集与目标蛋白结合的染色质进行分析。该技术自30年前第一次被提出以来,很少有所改进,很多科研工作者也因ChIP过程中出现的众多问题而深受困扰。直至最近Henikoff实验室提出CHIP新技术核酸酶靶向切割和释放(cleavage under target and release using nuclease,CUT&RUN),刷新了人们对DNA和蛋白研究方法的认识。
新技术刚产生,必然会引起大家的疑问,CUT&RUN也不例外:CUT&RUN是怎样工作的?有哪些优缺点?有没有相关的文献支持?有没有成品试剂盒?别着急,小编一一给您解答。
01 CUT&RUN是如何工作的?
首先将细胞固定在伴刀豆球蛋白A磁珠上,然后用洋地黄皂苷通透细胞膜,加入一抗和pAG-MNase(Protein A-Protein G-微球菌核酸酶),一抗募集pAG-MNase到靶蛋白上,0度条件下添加Ca2+激活pAG-MNase,并切割靶蛋白两侧的DNA,使其从基因组中释放出来,扩散到上清中。上清中的DNA纯化后进行qPCR或NG-Seq鉴定、定量。
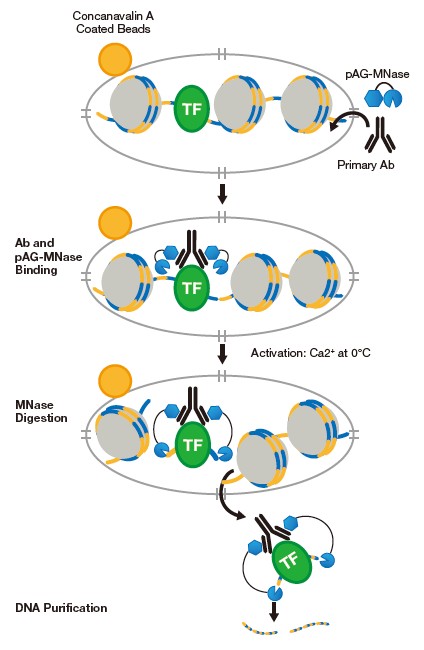
CUT&RUN原理图
△点击放大图片
02 两篇文献快速解析CUT&RUN的优点
小优通过以下两篇文献,从实验关键步骤、细胞量、测序结果信噪比等方面来解析此技术的优点。
文章一:Targeted in situ genome-wide profiling with high efficiency for low cell numbers

△点击放大图片
尽管随着技术的发展,ChIP-seq的改进可以实现TFs 6-8碱基对分辨率的映射,但实验结果还是存在很多问题,比如说ChIP的高背景限制了灵敏度,需要的细胞比较多以及交联和增溶产生的假象。
本文从流程上多方面验证了CUT&RUN相比于传统ChIP在细胞量和信噪比等方面的优势。
(1)在整个实验过程中,pMNase的量对片段化的结果至关重要
两个不同批次的滴定测试可以明显查出,研究者在500µl的体系中对600000个K562细胞加入不同浓度的pA-MN消化30分钟后,检测H3K27me3的基因组的荧光强度,结果可以看出CUT&RUN对pA-MN的浓度相对不敏感,其中将pA-MN的浓度提高到约100ng/ml以上,几乎没有其他释放。(图1)
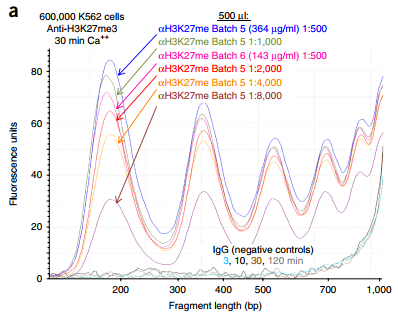
图1 两批样品500μl体积中,600,000个K562细胞,加入不同浓度的pA-MN,片段化之后,采用TapeStation 通过分析荧光强度来分析pA-MN对H3K27me3基因组片段化的影响。
△点击放大图片
(2)CUT&RUN技术用于少量细胞,信噪比很好
研究者检测了100-6000个K562细胞的H3K27me3的基因组分布,发现在低至100细胞时,CUT&RUN仍可以很好地检测出H3K27me3的峰,而且在异染色质区也一样可以看到峰。(图2)
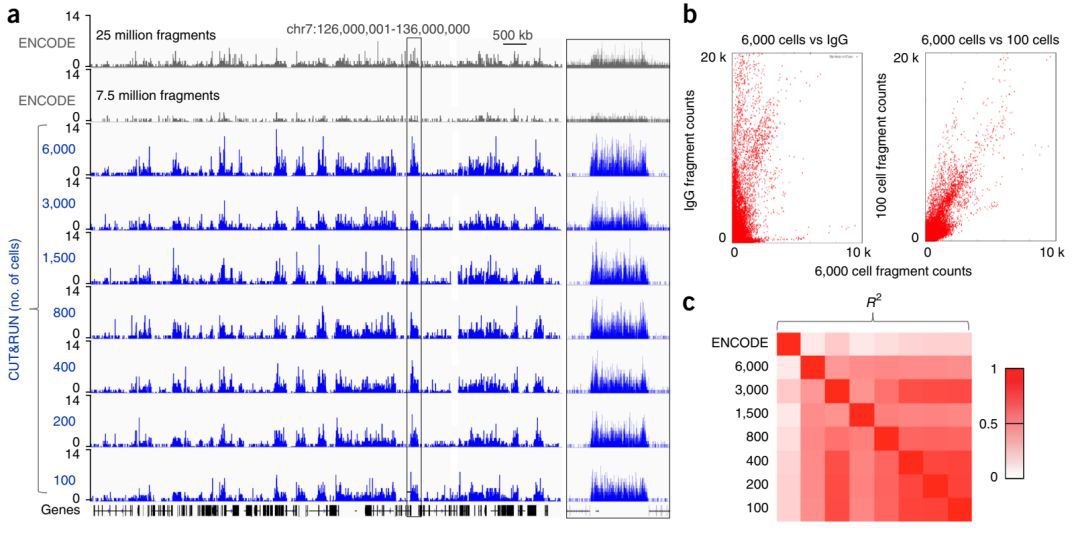
图2 CUT&RUN可以检测低至100细胞的H3K27me3基因组分布(a)ENCODE数据库中的K562细胞的H3K27me3 ChIP-seq结果,及6000-100个K562细胞的H3K27me3 CUT&RUN结果。(b)CUT&RUN结果的散点图,以50bp为一个bin,相对于阴性对照IgG,6000细胞与100细胞有明显更强的相关性。(c)热图显示6000-100细胞的CUT&RUN结果有很好的相关性。
△点击放大图片
(3)CUR&RUN技术检测的信噪比要比传统ChIP-seq高很多
研究者检测了转录因子CTCF的基因组结合位点,发现CUR&RUN技术检测比ENCODE的CTCF
ChIP-seq的信噪比要高很多,而且只需1.25万细胞就能得到很高质量的结果。尽管用1000细胞则会损失一些峰。(图3)
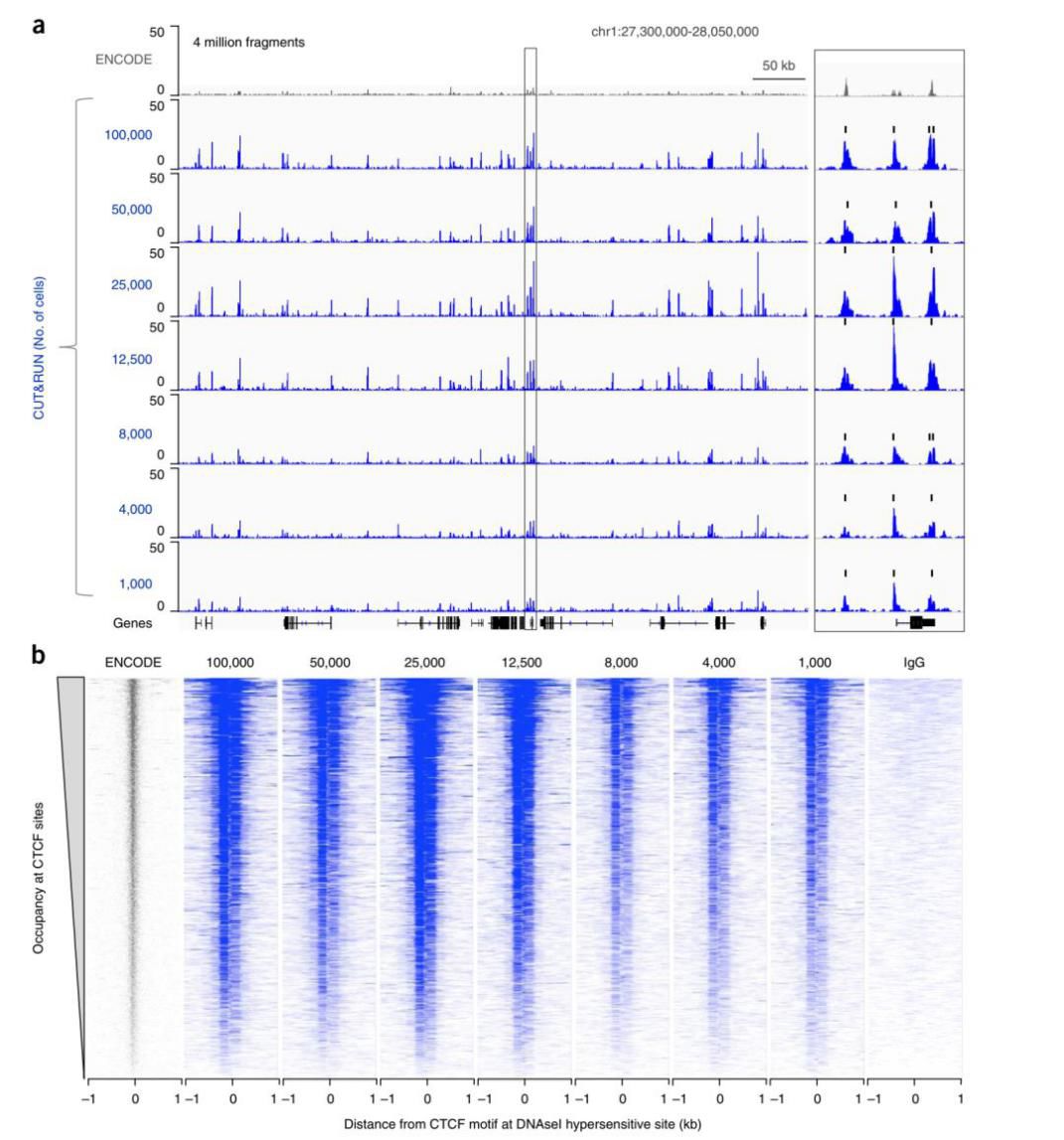
图3 用CUT&RUN检测CTCF的结合位点,比ENCODE中的CTCF ChIP-seq信噪比高很多。
△点击放大图片
※ 本文图2、图3分析源自CST公司。
文章二:An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites

△点击放大图片
为了验证CUT&RUN的效率,文章将CUT&RUN与CHIP进行比较,阐述了CUT&RUN的优点。与染色质免疫沉淀法(ChIP)将总染色质分离并溶解不同,CUT&RUN是在原位进行,可进行定量的高分辨率染色质定位和局部染色质环境的探测。当应用于酵母和人类细胞核时,CUT&RUN可产生精确的转录因子谱,同时避免交联和增溶问题。
(1)CUT&RUN产生限制消化的染色质片段
研究者选取了酿酒酵母组蛋白H2A、Abf1和Reb1转录因子,验证了在0度条件下,cut&run可以逐渐释放染色质片段首先是因为特异性抗体的存在;并说明了在ca2+存在的条件下,限制性剪切会立即发生。然后将上清中释放的片段化DNA和总DNA作对比,发现经CUT&RUN剪切后释放的片段化DNA完全可以覆盖转录因子结合位点。(图1)
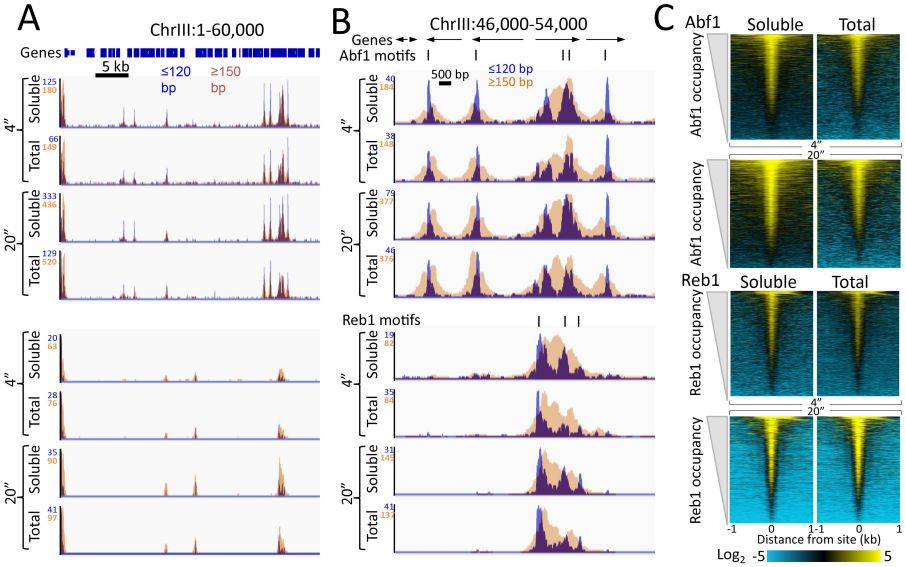
图1. 上清液部分(可溶性)与总DNA中Abf1和Reb1结合位点谱图比较
△点击放大图片
(2)CUT&RUN技术可以检测到转录因子的长距离3D结合的位点
研究者在K562中进行了CTCF的CUT&RUN和Native ChIP,发现Native ChIP只能检测到2298个峰。这些峰都有很高的CTCF motif分数,说明它们是CTCF直接结合的位点。而CUT&RUN却可以检测出约22000个峰,与X-ChIP的峰相似,包括高CTCF motif分数与低CTCF motif分数的峰,而且这些位点如果进行MNase-seq的话可以检测出峰,说明这些位点都有蛋白结合。以上说明CUT&RUN与X-ChIP一样可以检测到转录因子直接、间接结合的DNA。(图2)
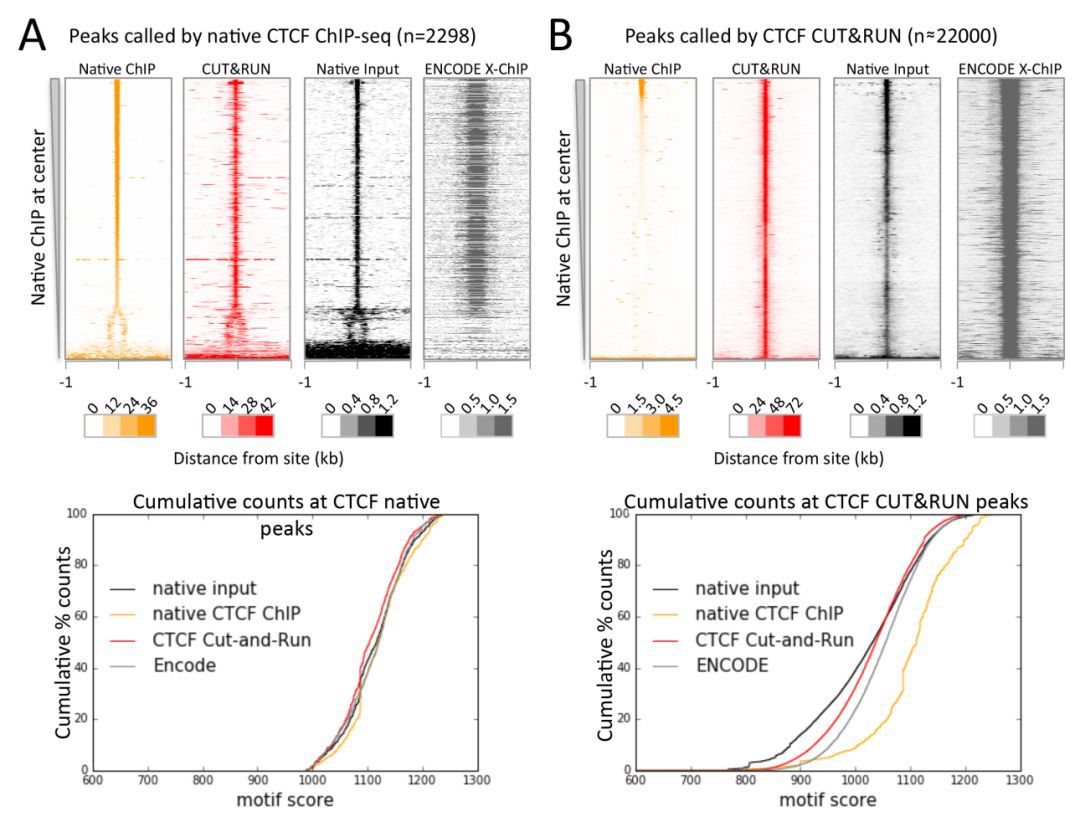
图2. CUT&RUN可以检测CTCF直接结合与间接结合的DNA位点。其中Native input即用MNase切割chromatin后进行测序的结果,有峰的地方说明有蛋白结合/保护
△点击放大图片
(3)CUT&RUN所需细胞量较少
经典的ChIP-seq实验需要大量细胞,而低细胞数ChIP仅限于丰富的蛋白质。研究者用60万到1000万K562细胞开展CTCF CUT&RUN实验。如果使用内部归一化,研究者观察到在消化过程中DNA剪切没有增加,但是通过对果蝇 spike-in DNA进行归一化,研究者发现DNA剪切水平随时间增加了约4倍。因此,CUT&RUN适用于蛋白质-DNA相互作用的准确定量。(图3)
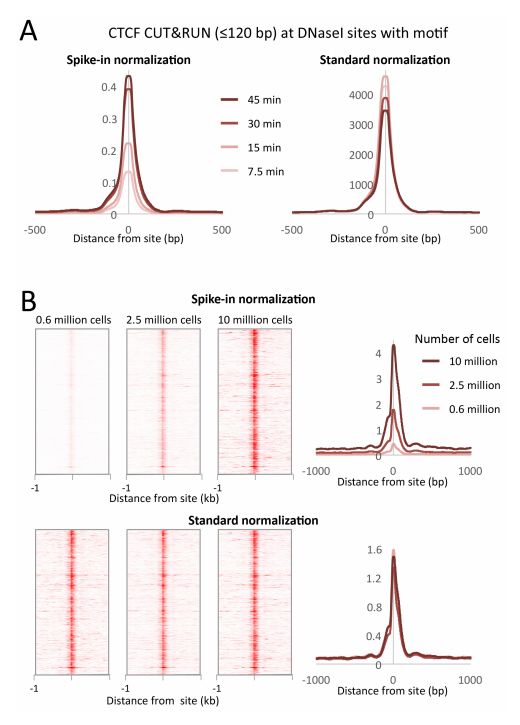
图3. CUT&RUN用于蛋白质-DNA相互作用简单定量。为了量化释放的片段,在裂解反应后添加1 ng果蝇DNA。
△点击放大图片
※ 本文图2分析源自CST公司。
看了上面两篇文章,发现CUT&RUN真是个好东西,具有诸多优势:所需细胞量少、背景低、易操作等等。稍安勿躁,小编这就带您来详细比较一下CUT&RUN(CST试剂盒货号#86652)技术与传统CHIP-Seq技术的优点,见表1。

表1. CUT&RUN和传统ChIP实验对比
△点击放大图片
03 CUT&RUN样本类型
此时的您是不是想立马买到这个盒子开展实验?别着急,还有一点,CST的CUT&RUN 试剂盒适用于什么样本呢?
CST的CUT&RUN适用于细胞样本。在开始实验前,建议您测试细胞对洋地黄皂苷的敏感性。不同细胞系对洋地黄皂苷细胞透化有不同的敏感性。虽然本试剂盒建议的洋地黄皂苷量应足以对大多数细胞系进行透化,但我们仍然建议对你的特定细胞系进行初步测试。
测试方法:收集细胞,用洋地黄皂苷通透细胞后进行台盼蓝染色。根据台盼蓝染色程度调整洋地黄皂苷的含量。充分通透会导致 > 90% 的细胞被台盼蓝染色。
04 CUT&RUN操作常见问题解决
原理、流程都已充分了解,或许您还是很担心使用CUT&RUN新产品实验结果不好,怎么办?没关系,小编整理出了一份实验过程中的Troubleshooting ,希望能帮大家预防和解决一些实验问题。

△点击放大图片
CUT&RUN毕竟是新技术,对于配套抗体的验证工作还在持续中,并不是所有的抗体我们都验证过,目前只有小部分抗体配合我们的试剂盒验证可使用,但是在我们的验证过程中,部分IF和ChIP应用的抗体可用于CUT&RUN,如您想自己验证,可作为参考。
05 更多相关文献
(1)Henikoff S , Skene P J . An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites[J]. eLife,6,(2017-01-06), 2017, 6.
(2)Peter J Skene, Jorja G Henikoff, Steven Henikoff. Targeted in situ genome-wide profiling with high efficiency for low cell numbers[J]. Nature Protocols, 2018, 13(5):1006-1019.
(3)Sarah, J, Hainer, et al. High-Resolution Chromatin Profiling Using CUT&RUN.[J]. Current protocols in molecular biology, 2019.
(4)Kasinathan S , Orsi G A , Zentner G E , et al. High-resolution mapping of transcription factor binding sites on native chromatin[J]. Nature Methods, 2013, 11(2):203-209.
(5)Meers M P , Bryson T D , Henikoff J G , et al. Improved CUT&RUN chromatin profiling tools[J]. eLife, 2019, 8.
(6)Meers M P , Janssens D H , Henikoff S . Pioneer Factor-Nucleosome Binding Events during Differentiation Are Motif Encoded[J]. Molecular cell, 2019.