如下操作步骤以产品货号180733的步骤为例,产品列表详见右下方推荐产品
实验步骤
- 1
- 2
- 3
- 4
-
从单细胞中扩增 Poly A+ mRNA
要从单细胞材料中扩增总 RNA,请使用“从单细胞中扩增总 RNA”的方案。对于纯化总 RNA 或富集 mRNA 的全转录组扩增,请使用“纯化 RNA 扩增”方案。
开始前的要点
· 该方案针对所有脊椎动物物种(例如,人类、小鼠、大鼠、分选细胞、组织培养细胞、显微镜下挑选的细胞或冷冻组织的显微切割细胞)的细胞(1-1000 个细胞)进行了优化。
· 该操作步骤不能用于细菌细胞。植物细胞或其他含有细胞壁的细胞和生物体也不适合。对于这些起始材料,首先纯化 RNA,然后使用“纯化 RNA 扩增”操作步骤进行 WTA。
· 该方案不能用于用福尔马林或其他交联剂处理的固定细胞(例如,通过激光显微切割从福尔马林固定、石蜡包埋的组织中获得的单细胞样本)。
· 使用 QIAseq FX Single Cell RNA Library Kit 的 1–1000 个完整细胞(例如人类或细胞培养细胞)的样本是全转录组扩增反应的最佳选择。避免在反应中使用超过 1000 个细胞,因为含有过多细胞的样品可能无法有效裂解。
· 通过使用单独的实验室设备(例如移液器、过滤移液器吸头、反应瓶等)避免试剂的任何 DNA 或 RNA 污染。在没有核酸的位置设置单细胞反应。
· 在无模板对照 (NTC) 中随机延伸引物(引物-多聚体形成)可能产生的高分子量 DNA 不包含遗传信息,不会影响下游应用的质量。在含有活细胞和足够 cDNA 的反应中,不会形成这些产物。
· 由于 QIAseq FX Single Cell RNA Library Kit 旨在从最少量的起始 RNA 中生成扩增的 cDNA,因此请务必采取适当的措施以避免无意中引入 RNase 污染。在使用 RNA 时,遵循适当的微生物学和无菌技术,创建和维护一个无 RNase 的环境。强烈建议使用刚打开的盒子或袋子中的一次性塑料管和移液器吸头。
· 用于全转录组扩增的试剂不适用于小 RNA 分子的扩增,例如 tRNA 或 miRNA。
· 请注意,最终反应体积为 59 μl。
· 尽管所有序列都得到了很好的表现,但扩增的 cDNA 并不包含全长 cDNA。扩增过程由随机引发的 cDNA 合成开始。因此,转录序列被分段扩增。由于连接反应的性质,DNA 片段可能不会按照它们最初存在于生物体中的顺序组装。然而,试剂盒化学旨在使这些事件变得罕见,因此核酸序列的检测和定量不受影响(例如,序列多态性、差异表达分析)。
开始前要做的事情
· 方案中描述的 Quantiscript RT 混合物、连接混合物和 REPLI-g SensiPhi 扩增混合物必须始终新鲜制备。它们不能储存以备后用。
· 所有缓冲液和试剂在使用前都应涡旋以确保充分混合。
· Quantiscript RT Enzyme Mix、Ligase Mix 和 REPLI-g SensiPhi DNA Polymerase 应在冰上解冻。所有其他组件都可以在室温 (15–25°C) 下解冻。
· 为了提高速度和便利性,操作步骤的所有孵育步骤都可以在热循环仪上进行预编程(表 1)。表 1. 热循环参数
步骤 时间 温度 附加评论 将所有步骤的加热盖设置为 50ºC 细胞裂解 5分钟 24ºC 添加裂解缓冲液(步骤 2) 3 分钟 95ºC ∞ 4ºC 抓住 gDNA 去除 10 分钟 42ºC 孵育前添加 gDNA Wipeout Buffer(步骤 4) ∞ 4°C 抓住 逆转录 60 分钟 42ºC 孵育前添加 Quantiscript RT 混合物(步骤 6) 3 分钟 95°C 停止逆转录 ∞ 4ºC 抓住 结扎 30分钟 24ºC 孵育前添加连接混合物(步骤 8) 5分钟 95°C 停止连接 ∞ 4ºC 抓住 全转录组放大 2 h 30ºC 孵化前添加 REPLI-g SensiPhi 扩增混合物(步骤 10) 5分钟 65ºC 灭活所有酶 ∞ 4ºC 冷却扩增的 cDNA
程序
1. 将 7 μl 细胞材料(与 PBS 一起提供)放入微量离心管中。如果使用少于 7 μl 的细胞材料,添加 H20 sc 使体积达到 7 μl。
注意:立即进行第 2 步。
2. 添加 4 μl 裂解缓冲液。轻轻轻弹试管小心混合,然后短暂离心。
注意:确保细胞材料不会粘在半月板上方的管壁上,并且裂解缓冲液与细胞材料的混合是完全的。
3. 在 24°C 下孵育 5 分钟,然后在 95°C 下孵育 3 分钟。冷却至 4°C。
4. 加入 2 μl gDNA Wipeout Buffer,涡旋混合并短暂离心。
5. 在 42°C 下孵育 10 分钟。如果需要更多时间来准备下一步,请放在冰上。
6. 准备 Quantiscript RT 组合(表 2)。向裂解的细胞样品中加入 6 μl Quantiscript RT Mix,涡旋混合并短暂离心。
注意:Quantiscript RT 混合物必须新鲜制备。表 2. Quantiscript RT 混合物的制备*
零件 体积/反应 RT/聚合酶缓冲液 4微升 寡核苷酸引物 1微升 Quantiscript RT 酶混合物 1微升 总音量† 6微升 * 要为多个反应制备 Quantiscript RT 混合液,请根据反应数量扩大规模。
通过涡旋和短暂离心混合。
7. 在 42ºC 下孵育 60 分钟。通过在 95ºC 下孵育 3 分钟来停止反应,然后在冰上冷却。
8. 准备结扎组合(表 3)。将 10 μl 连接混合物添加到步骤 7 的 RT 反应中。通过涡旋和短暂离心混合。
重要提示:制备连接混合物时,请按表 3 中所示的顺序添加组分。
注意:连接混合物必须新鲜制备。表 3. 连接混合物的制备*
零件 体积/反应 连接酶缓冲液 8微升 连接酶混合物 2微升 总音量† 10微升 * 要为多个反应制备连接混合物,请根据反应次数放大。
通过涡旋和短暂离心混合。
9. 在 24ºC 下孵育 30 分钟。通过在 95°C 下孵育 5 分钟来停止反应,然后在冰上冷却。
10. 准备 REPLI-g SensiPhi 放大组合(表 4)。将 30 μl REPLI-g SensiPhi 扩增混合物添加到步骤 9 的连接反应中。通过涡旋和短暂离心混合。
注意:REPLI-g SensiPhi 扩增混合物必须新鲜制备。表 4. REPLI-g SensiPhi 扩增混合物的制备*
零件 体积/反应 REPLI-g sc 反应缓冲液 29微升 REPLI-g SensiPhi DNA 聚合酶 1微升 总音量† 30微升 * 要为多个反应制备 REPLI-g SensiPhi 扩增混合物,请根据反应数量进行放大。
通过涡旋和短暂离心混合。
11. 在 30ºC 下孵育 2 小时。
12. 通过在 65ºC 下孵育 5 分钟来停止反应,然后在冰上冷却。
13. 如果不直接使用,将扩增的 cDNA 储存在 –15ºC 至 –30ºC 直至下游应用需要。我们建议以 100 ng/μl 的最低浓度储存扩增的 DNA。
注意:在无模板对照(NTC)中通过引物随机延伸(引物-多聚体形成)产生的高分子量 DNA 不包含遗传信息,不会影响下游应用的质量。这些产品被 WTA 期间存在的活细胞的 cDNA 竞争。
14. 扩增后的 cDNA 可直接用于文库构建,也可用于靶向扩增和文库构建。扩增的 cDNA 与纯化的基因组 DNA 相似,长度约为 2,000–70,000 bp。
注意:如果需要对扩增的 cDNA 进行量化,请按照附录 B 中的说明进行操作。光密度 (OD) 测量会高估第 12 步中的扩增 cDNA,因此不应使用。附录内容可在QIAGEN官网下载货号为180733的完整说明书进行了解。
-
从单细胞中扩增总 RNA
该方案用于从单细胞材料中扩增总 RNA。请注意,rRNA 也使用此操作步骤进行放大,并且在放大后将代表所有 cDNA 的高百分比,因此在结果数据集中读取的百分比很高。为了富集包括 mRNA 在内的多聚腺苷酸化 RNA,我们建议使用“从单细胞中扩增 Poly A+ mRNA”的方案,该方案可避免 rRNA 的扩增并生成非常适合 NGS 的 cDNA。对于纯化 RNA 的全转录组扩增,请参阅“纯化 RNA 的扩增”方案。
开始前的要点
· 该方案针对所有脊椎动物物种(例如,人类、小鼠、大鼠、分选细胞、组织培养细胞、显微镜下挑选的细胞或冷冻组织的显微切割细胞)的细胞(1-1000 个细胞)进行了优化。
· 该操作步骤不能用于细菌细胞。植物细胞或其他含有细胞壁的细胞也不适合。对于这些起始材料,首先纯化 RNA,然后使用“纯化 RNA 扩增”操作步骤进行 WTA。
· 该方案不能用于用福尔马林或其他交联剂处理的固定细胞(例如,通过激光显微切割从福尔马林固定、石蜡包埋的组织中获得的单细胞样本)。
· 使用 QIAseq FX 单细胞 RNA 文库试剂盒,1–1000 个完整细胞(例如人类或细胞培养细胞)的样本是全转录组扩增和后续文库制备的最佳选择。避免在反应中使用超过 1000 个样品,因为含有过多细胞的样品可能无法有效裂解。
· 通过使用单独的实验室设备(例如移液器、过滤移液器吸头、反应瓶等)避免试剂的任何 DNA 或 RNA 污染。在没有核酸的位置设置 REPLI-g 单细胞反应。
· 在无模板对照 (NTC) 中随机延伸引物(引物-多聚体形成)产生的高分子量 DNA 不包含遗传信息,不会影响下游应用的质量。这些产品被 WTA 期间存在的活细胞 DNA 所取代。
· 由于 QIAseq FX Single Cell RNA Library Kit 旨在从最少量的起始 RNA 中生成扩增的 cDNA,因此请采取适当的措施以避免无意中引入 RNase 污染。通过遵循适当的微生物学和无菌技术,在处理 RNA 时创建和维持无 RNase 的环境。强烈建议使用刚打开的盒子或袋子中的一次性塑料管和移液器吸头。
· 用于全转录组扩增的试剂不适用于小 RNA 分子的扩增,例如 tRNA 或 miRNA。
开始前要做的事情
· 方案中描述的 Quantiscript RT 混合物、连接混合物和 REPLI-g SensiPhi 扩增混合物必须始终新鲜制备。它们不能储存以备后用。
· 所有缓冲液和试剂在使用前都应涡旋以确保充分混合。
· Quantiscript RT Enzyme Mix、Ligase Mix 和 REPLI-g SensiPhi DNA Polymerase 应在冰上解冻。所有其他组件都可以在室温 (15–25°C) 下解冻。
· 为了提高速度和便利性,操作步骤的所有孵育步骤都可以在热循环仪上进行预编程(表 5)。表 5. 热循环参数
步 时间 温度 附加评论 将所有步骤的加热盖设置为 50ºC 细胞裂解 5分钟 24ºC 添加裂解缓冲液(步骤 2) 3 分钟 95ºC ∞ 4ºC 抓住 gDNA 去除 10 分钟 42ºC 孵育前添加 gDNA Wipeout Buffer(步骤 4) ∞ 4°C 抓住 逆转录 60 分钟 42ºC 孵育前添加 Quantiscript RT 混合物(步骤 6) 3 分钟 95°C 停止逆转录 ∞ 4ºC 抓住 结扎 30分钟 24ºC 孵育前添加连接混合物(步骤 8) 5分钟 95°C 停止连接 ∞ 4ºC 抓住 全转录组放大 2 h 30ºC 孵化前添加 REPLI-g SensiPhi 扩增混合物(步骤 10) 5分钟 65ºC 灭活所有酶 ∞ 4ºC 抓住
程序
1. 将 7 μl 细胞材料(与 PBS 一起提供)放入微量离心管中。如果使用少于 7 μl 的细胞材料,添加 H20 sc 使体积达到 7 μl。
2. 添加 4 μl 裂解缓冲液。轻轻轻弹试管小心混合,然后短暂离心。
注意:确保细胞材料不会粘在半月板上方的管壁上。
3. 在 24°C 下孵育 5 分钟,然后在 95°C 下孵育 3 分钟。冷却至 4°C。
4. 加入 2 μl gDNA Wipeout Buffer,涡旋混合并短暂离心。
5. 在 42°C 下孵育 10 分钟。如果需要更多时间来准备下一步,请放在冰上。
6. 准备 Quantiscript RT 组合(表 6)。向裂解的细胞样品中加入 7 μl Quantiscript RT mix,涡旋混合并短暂离心。
注意:Quantiscript RT 混合物必须新鲜制备。表 6. Quantiscript RT 混合物的制备*
零件 体积/反应 RT/聚合酶缓冲液 4微升 随机引物 1微升 寡核苷酸引物 1微升 Quantiscript RT 酶混合物 1微升 总音量† 7微升 * 要为多个反应制备 Quantiscript RT 混合液,请根据反应数量扩大规模。
通过涡旋和短暂离心混合。
7. 在 42ºC 下孵育 60 分钟。通过在 95ºC 下孵育 3 分钟来停止反应,然后在冰上冷却。
8. 准备结扎混合物(表 7)。将 10 μl 连接混合物添加到步骤 7 的 RT 反应中。通过涡旋和短暂离心混合。
重要提示:制备连接混合物时,请按表 7 中所示的顺序添加组分。
注意:连接混合物必须新鲜制备。表 7. 连接混合物的制备*
零件 体积/反应 连接酶缓冲液 8微升 连接酶混合物 2微升 总音量† 10微升 * 要为多个反应制备连接混合物,请根据反应次数放大。
通过涡旋和短暂离心混合。
9. 在 24ºC 下孵育 30 分钟。通过在 95°C 下孵育 5 分钟来停止反应,然后在冰上冷却。
10. 准备 REPLI-g SensiPhi 放大组合(表 8)。将 30 μl REPLI-g SensiPhi 扩增混合物添加到步骤 9 的连接反应中。通过涡旋和短暂离心混合。
注意:REPLI-g SensiPhi 扩增混合物必须新鲜制备。表 8. REPLI-g SensiPhi 扩增混合物的制备*
零件 体积/反应 REPLI-g sc 反应缓冲液 29微升 REPLI-g SensiPhi DNA 聚合酶 1微升 总音量† 30微升 * 要为多个反应制备 REPLI-g SensiPhi 扩增混合物,请根据反应数量进行放大。
通过涡旋和短暂离心混合。
11. 在 30ºC 下孵育 2 小时。
12. 通过在 65ºC 下孵育 5 分钟来停止反应,然后在冰上冷却。
13. 如果不直接使用,将扩增的 cDNA 储存在 –15 ºC 至 –30ºC 直至下游应用需要。我们建议以 100 ng/μl 的最低浓度储存扩增的 cDNA。
注意:在无模板对照(NTC)中通过引物随机延伸(引物-多聚体形成)产生的高分子量 DNA 不包含遗传信息,不会影响下游应用的质量。这些产物被 MDA 期间存在的活细胞产生的 cDNA 竞争。
14. 扩增后的 cDNA 可直接用于文库构建,也可用于靶向扩增和文库构建。扩增的 cDNA 与纯化的基因组 DNA 相似,长度约为 2000–70,000 bp。
注意:如果需要对扩增的 cDNA 进行量化,请按照附录 B 中的说明进行操作。光密度 (OD) 测量会高估第 12 步中的扩增 DNA,因此不应使用。 -
纯化 RNA 的扩增
开始前的要点
· 该方案可应用于任何类型的纯化 RNA,例如总 RNA、poly A+ RNA(使用 GeneRead Pure mRNA 试剂盒)或 rRNA 耗尽的 RNA(使用 GeneRead rRNA Depletion 试剂盒)。它不适用于降解的 RNA,例如来自 FFPE 组织的 RNA。
· 用于纯化 RNA 的 WTA 的具体方案取决于起始材料和下游应用。
· 使用 50 pg – 100 ng 的纯化 RNA 进行 WTA 实验。
· 通过使用单独的实验室设备(例如移液器、过滤移液器吸头、反应瓶等)避免试剂的任何 DNA 或 RNA 污染。在没有核酸的位置设置 REPLI-g 单细胞反应。
· 在无模板对照 (NTC) 中随机延伸引物(引物-多聚体形成)产生的高分子量 DNA 不包含遗传信息,不会影响下游应用的质量。这些产品被 WTA 期间存在的活细胞 DNA 所取代。
· 由于 QIAseq FX Single Cell RNA Library Kit 旨在从最少量的起始 RNA 中生成扩增的 cDNA,因此请采取适当的措施以避免无意中引入 RNase 污染。通过遵循适当的微生物学和无菌技术,在处理 RNA 时创建和维持无 RNase 的环境。强烈建议使用刚打开的盒子或袋子中的一次性塑料管和移液器吸头。
· 用于全转录组扩增的试剂不适用于小 RNA 分子的扩增,例如 tRNA 或 miRNA。
· 尽管所有序列都得到了很好的表现,但扩增的 cDNA 并不包含全长 cDNA。扩增过程由随机引物和寡 dT - 引物 cDNA 合成开始。因此,转录序列被分段扩增。由于连接反应的性质,DNA 片段可能不会按照它们最初存在于生物体中的顺序组装。然而,试剂盒化学旨在使这些事件变得罕见,因此在下游 NGS 应用中核酸序列的检测不受影响(例如,多态性)。
开始前要做的事情
· 方案中描述的 Quantiscript RT 混合物、连接混合物和 REPLI-g SensiPhi 扩增混合物必须始终新鲜制备。它们不能储存以备后用。
· 所有缓冲液和试剂在使用前都应涡旋以确保充分混合。
· Quantiscript RT Enzyme Mix、Ligase Mix 和 REPLI-g SensiPhi DNA Polymerase 应在冰上解冻。所有其他组件都可以在室温 (15–25°C) 下解冻。
· 为了提高速度和便利性,操作步骤的所有孵育步骤都可以在热循环仪上进行预编程。使用与起始材料对应的操作步骤中列出的循环参数。
程序
1. 将 8 μl 纯化的 RNA (>50 pg) 放入微量离心管中。如果使用少于 8 μl 的纯化 RNA,添加 H20 sc 使体积达到 8 μl。
2. 加入 3 μl NA 变性缓冲液,涡旋混合并短暂离心。
3. 在 95°C 下孵育 3 分钟,然后冷却至 4°C。
4. 继续执行“从单细胞扩增 Poly A+ mRNA”或“从单细胞扩增总 RNA”操作步骤的第 4 步。 -
使用 QIAseq FX 单细胞扩增 cDNA 进行酶促片段化和文库制备
开始前的要点
· 该操作步骤用于使用 QIAseq FX Single Cell DNA Library Kit 为 Illumina NGS 平台构建测序文库。
· 该方案还需要以下 QIAGEN 产品: 对于反应净化和文库构建后接头二聚体的去除,Agencourt AMPure XP Beads(货号 A63880、A63881)或 GeneRead Size Selection Kit(货号 180514)是需要,应单独订购。
· 扩增的 cDNA 在开始前应在 H2O 中稀释。
开始前要做的事情
· 程序周期。为了提高速度和便利性,操作步骤的所有孵育步骤都可以预先编程并保存在热循环仪上(表 5)。
请参阅表 5 以确定将输入 DNA 片段化为所需大小所需的时间和方案。
· 准备新鲜的 80% 乙醇。
· 制备缓冲液 10 mM Tris-HCl,pH 8.0
· 程序热循环仪。为了提高速度和便利性,操作步骤的所有孵化步骤都可以预先编程和保存。
程序:酶裂解和文库制备
FX 单管碎裂、末端修复和A-加法
1. 在冰上解冻所有套件组件。一旦试剂解冻,通过快速涡旋彻底混合缓冲液以避免任何局部浓度。使用前短暂旋转涡旋试剂。根据表 9 对热循环仪进行编程并启动程序。如果可能,将加热盖的温度设置为 ~70°C。
2. 当热循环仪模块达到 4°C 时,暂停程序。表 9. 扩增的 cDNA 片段化反应条件
步 温度 孵育时间(片段大小 300 bp) 孵育时间(片段大小 500 bp) 1 4°C 1分钟 1分钟 2 32°C 15 分钟* 10 分钟 3 65°C 30分钟 30分钟 4 4°C 抓住 抓住 *已完成文库的插入片段大小由步骤 2 的持续时间决定。使用 200–1000 ng 输入 DNA,如果使用 FX 增强剂,15 分钟的片段化时间会产生约 300 bp 的片段分布,如果使用此组件,则为 500 bp被省略。可以通过改变第 2 步的持续时间来调整片段大小。使用带加热盖的热循环仪。
3. 在 H2O sc 中稀释扩增的 cDNA 1:3。这应该在 10 μl H2O sc (50-100 ng/μl) 中得到 500 – 1000 ng 总扩增 DNA。如果您已对 cDNA 进行了定量,则在 FX 反应中输入的未稀释 cDNA 不得超过 5 μl。在 PCR 管或条带中吸取 10 μl 稀释的 DNA,并将它们放在冰或冷却块上。
4. 如果所需的库片段大小为 300 bp,则根据表 10 在冰上制备 FX 反应混合物,或根据表 11 制备 500 bp 的库片段大小并通过移液混合。按照表中所述的相同顺序添加 FX Reaction Mix 的组分。在添加 FX 酶混合物之前,用移液器上下移液器混合物。您可以根据处理的样本数量扩大 FX Reaction Mix。表 10. 300 bp 插入片段大小的 FX 反应设置
零件 体积/反应* FX 缓冲器,10x 5微升 水蒸汽 20微升 效果增强器 5微升 FX 酶混合物 10微升 总反应体积 40微升 * 通过移液器混合,并保持在冰上。
表 11. 500 bp 插入片段大小的 FX 反应设置
零件 体积/反应* FX 缓冲器,10x 5微升 水蒸汽 25微升 FX 酶混合物 10微升 总反应体积 40微升 * 通过移液器混合,并保持在冰上。
5. 将 40 μl FX Reaction Mix 添加到冰上每个稀释的扩增 cDNA 样品中,轻轻涡旋混合。
6. 短暂旋转 PCR 板/管,立即转移到预冷的热循环仪 (4°C) 并恢复程序。碎片程序完成后,将样品转移到冰上。
7. 立即按照下一个操作步骤中的说明进行适配器连接。
适配器连接
8. 使用前在室温下平衡 Agencourt AMPure XP 微珠 20-30 分钟。
9. 涡旋并向下旋转适配器板。取下保护性适配器板盖,小心地刺穿箔密封并将 5 μl 从一个 DNA 适配器孔转移到前一个方案中的每个 50 μl 样品中。跟踪用于每个样品的条形码。
10. 更换适配器板盖并冷冻未使用的适配器。转接板在至少 10 次冻融循环中是稳定的。
重要提示:每个连接反应只能使用一个接头。如果使用其他供应商的适配器,请遵循制造商的说明。
11. 根据表 12 在冰上的单独试管中制备连接预混液(每个 DNA 样品)。充分混合或在低转速下轻轻涡旋。表 12. 连接预混液(每个样品)
零件 体积/反应* DNA 连接酶缓冲液,5x 20微升 H2Osc 15微升 DNA连接酶 10微升 总反应体积 45微升 * 通过移液器混合,并保持在冰上。
12. 在每个样品中加入 45 μl 的连接预混液。充分混合并在 20°C 下孵育 15 分钟。
重要提示:请勿使用带加热盖的热循环仪。
13. 使用 0.8x (80 μl) Agencourt AMPureXP 珠子立即进行接头连接清理(步骤 14–23)。
14. 将 80 μl 重悬的 Agencourt AMPure XP 珠浆添加到每个连接的样品中,并通过移液器或轻轻涡旋充分混合。
15. 在室温下孵育混合物 5 分钟。
16. 将磁珠放在磁性支架上 2 分钟,然后小心丢弃上清液。
17. 通过向每个颗粒中加入 200 μl 新鲜的 80% 乙醇来清洗磁珠。将磁珠放在磁力架上 2 分钟,然后小心丢弃上清液。
18. 重复洗涤步骤 17 一次,总共进行 2 次乙醇洗涤。
19. 在磁力架上孵育 5-10 分钟或直到珠子变干。避免过度干燥,这可能会导致 DNA 回收率降低。从磁性支架上取下。
20. 通过重悬于 52.5 μl 10 mM Tris-HCl,pH 8.0 中进行洗脱。磁力架上的颗粒珠。小心地将 50 μl 上清液转移到新的 PCR 板上。
21. 进行第二次纯化。向每个样品中加入 50 μl 重悬的 1x Agencourt AMPure XP 微珠并混合。
22. 按照步骤 15–19。
23. 通过重悬于 26 μl 10 mM Tris-HCl,pH 8.0 中进行洗脱。将磁珠放在磁力架上。小心地将 23.5 μl 上清液转移到新的 PCR 板中。将纯化的文库储存在 –20°C 直至准备好进行测序。
24. 使用毛细管电泳装置或其他类似方法评估库的质量。检查库片段的正确大小分布(图 2)以及是否缺少适配器或适配器二聚体。
注意:DNA 片段的中值大小应根据与文库片段连接的接头的大小(例如,对于 GeneRead Adapter I Set 1-plex 或 GeneRead Adapter I Set 12-plex,添加 120 bp )。
注意:中间片段大小可用于后续基于 qPCR 的量化方法。这个中值大小可以在扩增文库和无 PCR 文库之间移动大约 100 倍。 30 个基点。
图 2. 生成库的毛细管电泳设备轨迹。毛细管电泳设备跟踪数据显示 4 个复制完成的文库的正确大小分布以及不存在接头或接头二聚体。△点击放大图片
25. 使用 QIAseq Library Quant Assay Kit(产品编号 333314,单独出售)或其他类似方法对文库进行量化。
注意:强烈建议通过 qPCR 进行文库定量,以确保准确的文库稀释和聚类,最大限度地提高通过过滤器读取。毛细管电泳或 Qubit®测量可能会高估文库数量,因为它们无法区分可测序文库片段与仅包含一个接头的插入片段。
使用 200 ng – 1 μg WTA cDNA 输入,应生成足够的文库用于在 Illumina 平台上进行测序,而无需进一步 PCR 扩增(图 3)。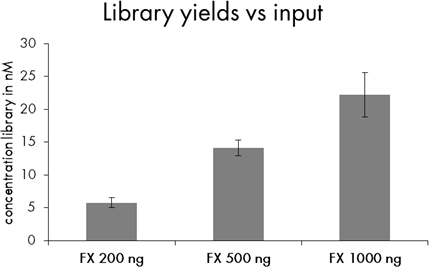
图 3:文库产量与 WTA DNA 输入的对比。绘制的数据是用 SD 进行三次反应的手段。△点击放大图片
26. 纯化后的文库可在 –20°C 下安全储存,直至需要。


