如下操作步骤以产品货号333955的步骤为例,产品列表详见右下方推荐产品
实验步骤
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
-
核酸片段化,标准样品
开始前的要点
· 该操作步骤描述了来自“标准样品”(即细胞或组织)的核酸片段化。 FFPE样本 的碎片化,请参考“操作步骤:核酸碎片,FFPE 样品”.
· 该方案设计用于使用总核酸洗脱液(含有 DNA+RNA 的洗脱液)或单独的 DNA 和 RNA 洗脱液。
· 在执行“方案:在单管中结合靶向 DNA+RNA 富集”时,建议的 DNA 量为 10–40 ng 。
· 在单独的试管中分离靶向 DNA 和 RNA 富集”时,建议的 DNA 量为 20-80 ng 。
· 推荐的 RNA 量为 10 ng 至 250 ng 总 RNA 。在处理总核酸样本时,我们建议根据定量 DNA 的量输入。
· 在冰上建立反应。
· 不要涡旋任何试剂或反应。
程序
1. 在冰上解冻核酸样本。轻轻混合,短暂离心以收集管壁上的残留液体,然后放回冰上。
2. 准备碎片所需的试剂。
2a.在室温 (15–25°C) 下解冻碎片缓冲液、 10x 和 FERA 溶液。
2b.轻弹试管混合,然后短暂离心。
注意: 副反应减少剂和碎裂酶混合物应在使用前从冰箱中取出并置于冰上。使用后,立即将酶放回冰箱。
3. 在冰上,根据 表7 。短暂离心,上下吹打混合 10-12 次 ,然后再次短暂离心。
注意: 如果设置了多个反应,则准备的预混液体积比反应总数所需的量大 10% 。表 7. 核酸片段化反应混合物
零件 体积/反应 DNA(参见“要点在开始之前“ 部分)* 变量 A DNA(参见“要点在开始之前“ 部分)* 变量 B 碎片缓冲液,10x 2µl FERA 溶液 0.6µl 副反应还原剂 1.6 µl 碎片酶混合物 4µl 无核酸酶水 11.8 µl – 可变 A (DNA) – 可变 B (RNA) 全部的 20µl *可以添加同时包含 DNA 和 RNA 的总核酸,而不是单独添加 DNA 和 RNA 。如果添加总核酸,则输入量基于 DNA。
4. 根据表 8。使用仪器的加热盖。表 8. 核酸片段化的孵育条件
步 孵化温度 孵化时间 1 4°C 1分钟 2 32°C 24 分钟 3 72°C 30分钟 4 4°C 抓住
5. 在将管/板添加到热循环仪之前,启动程序。当热循环仪达到 4°C 时,暂停程序。
重要提示: 热循环仪必须预冷并在 4°C 下暂停。
6. 转移步骤中准备的 管/板3 到预冷的热循环仪并恢复程序。
7. 完成后,让热循环仪回到 4°C 。
8. 将样品放在冰上并立即进行“方案: RNA多聚腺苷酸化”. -
核酸片段化,FFPE 样品
开始前的要点
· 该操作步骤描述了 FFPE 样品中核酸的片段化。关于“标准样品”(即细胞或组织)的碎片化,请参阅“操作步骤: 核酸片段化,标准样品”。
· 该方案设计用于使用总核酸洗脱液(含有 DNA+RNA 的洗脱液)或单独的 DNA 和 RNA 洗脱液。
· 如果使用了 QIAseq QuantiMIZE 试剂盒,建议的 FFPE DNA 量最高为 250 ng DNA如果使用替代方法来确定 FFPE DNA 的浓度,则最多可以使用 100 ng DNA。建议的 FFPE RNA 量为 250 ng 总 RNA(对于“严重”片段化的 FFPE 样品最高为 500 ng,“严重”被定义为具有少于 40% 片段的样品 >200 nt 在生物分析仪上进行涂片分析) .在处理总核酸样本时,我们建议根据定量 DNA 的量输入。
· 在冰上建立反应。
· 不要涡旋任何试剂或反应。
程序
1. 在冰上解冻核酸样本。轻轻混合,短暂离心以收集管壁上的残留液体,然后放回冰上。
2. 准备碎片所需的试剂。
2a.在室温下解冻碎片缓冲液、10x 和 FERA 溶液。2b.轻弹试管进行混合,然后短暂离心。
注意:副反应还原剂和碎裂酶混合物应从在使用前将冰箱放在冰上。使用后,立即将酶放回冰箱。
3. 在冰上,根据表 9。短暂离心,上下吹打混合 10-12 次,然后再次短暂离心。
注意:如果设置了多个反应,请准备体积大于 10% 的预混液比反应总数所需的数量。表 9. 核酸片段化反应混合物
零件 体积/反应 DNA(参见“要点在开始之前“ 部分)* 变量 A RNA(参见“要点在开始之前“ 部分)* 变量 B 碎片缓冲液,10x 2µl FERA 溶液 0.6µl 副反应还原剂 1.6µl 无核酸酶水 11.8 µl – 可变 A (DNA) – 可变 B (RNA) 全部的 16µl *可以添加同时包含 DNA 和 RNA 的总核酸,而不是单独添加 DNA 和 RNA。如果添加总核酸,则输入量基于 DNA。
4. 在 37°C 下孵育 15 分钟,然后置于冰上。
5. 在每个反应中加入 4 µl Fragmentation Enzyme Mix。短暂离心,上下吹打混合 10-12 次(不要涡旋),然后再次短暂离心。
重要提示:在整个反应设置过程中,将反应管/反应板置于冰上。
6.根据表 10。使用仪器的加热盖。表 10. 核酸片段化的孵育条件
步 孵化温度 孵化时间 1 4°C 1分钟 2 32°C 14 分钟 3 72°C 30分钟 4 4°C 抓住
7. 在将管/板添加到热循环仪之前,启动程序。当热循环仪达到 4°C 时,暂停程序。
重要提示:热循环仪必须预冷并在 4°C 下暂停。
8. 将步骤 5 中准备的管/板转移到预冷的热循环仪并恢复程序。
9. 完成后,让热循环仪回到 4°C。
10. 将样品置于冰上,立即进行“方案:RNA 多聚腺苷酸化”. -
RNA 多聚腺苷酸化
开始前的要点
· 产品来自“操作步骤:核酸片段化,标准样品”, 或者 ”方案:核酸片段化,FFPE 样品”, 是该操作步骤的起始材料。
· 在冰上建立反应。
· 不要涡旋任何试剂或反应。
程序
1. 准备聚腺苷酸化所需的试剂。
1a.在室温下解冻 PAP 稀释缓冲液、10x 和 ATP 溶液。1b.轻弹试管混合,然后短暂离心。
注意:T4 多核苷酸激酶和 PAP 酶应在使用前从冰箱中取出并置于冰上。使用后,立即将酶放回冰箱。
2. 用 18 µl 无核酸酶水稀释 2 µl 10x PAP 稀释缓冲液,制备 1x PAP 稀释缓冲液。
3. 使用 1x PAP 稀释缓冲液将等分的 PAP 酶从 5 U/µl 稀释至2 U/微升。短暂离心,上下吹打混合 10-12 次,然后再次短暂离心。
4. 根据准备 RNA 多腺苷酸化混合物表 11。短暂离心,上下吹打混合 10-12 次,然后再次短暂离心。
注意:如果设置了多个反应,则准备的预混液体积比反应总数所需的量大 10%。表 11. RNA 多聚腺苷酸化反应混合物
零件 体积/反应 裂解反应(已在试管中) 20µl ATP 溶液 1.25µl T4 多核苷酸激酶 1µl 稀释的 PAP 酶 (2 U/µl)* 1 µl 无核酸酶水 1.75µl 全部的 25µl *确保使用 1x PAP 稀释缓冲液将 PAP 酶从其原液 5U/µl 浓度稀释到 2U/µl。
5. 根据以下步骤在热循环仪中孵育反应表 12。使用仪器的加热盖。表 12. RNA 多聚腺苷酸化的孵育条件
步 孵化温度 孵化时间 1 4°C 1分钟 2 30°C 10 分钟 3 4°C 抓住
6. 完成后,将反应置于冰上并继续“方案:DNA 连接”. -
DNA 连接
开始前的要点
· “方案:RNA 多聚腺苷酸化“, 是该操作步骤的起始材料。
· 在冰上建立反应。
· 不要涡旋任何试剂或反应。
· QIAseq Beads 用于所有反应净化。
· 重要提示:每天准备新鲜的 80% 乙醇。
· 确保 QIAseq Beads 始终完全混合。这需要快速工作并在使用前立即重新悬浮珠子。如果操作步骤出现延迟,只需涡旋磁珠即可。
程序
1. 准备 DNA 连接所需的试剂。
1a.解冻 DNA 连接适配器;连接缓冲液,5x;和室温下的连接溶液。
1b.轻弹试管混合,然后短暂离心。
注意:DNA 连接酶应在使用前从冰箱中取出并置于冰上。使用后,立即将酶放回冰箱。
2. 根据制备 DNA 结扎混合物表 13。短暂离心,上下吹打混合 10-12 次,然后再次短暂离心。
重要提示:移液器缓慢混合。反应混合物非常粘稠。不要涡旋。
注意:如果设置了多个反应,则准备的预混液体积比反应总数所需的量大 10%。表 13. DNA 连接反应混合物
零件 体积/反应 RNA 多聚腺苷酸化反应(已在试管中) 25µl 连接缓冲液,5x 10µl DNA 连接接头 2.8 µl DNA 连接酶 5µl 连接溶液* 7.2µl 全部的 50µl *连接溶液非常粘稠。它应该单独添加到每个反应中,而不是与其他组分预混合作为主混合物。不要用连接溶液涂抹移液器吸头的外部,因为这样做可能会增加多余的体积。
3. 根据以下步骤在热循环仪中孵育反应表 14。
重要提示:请勿使用加热的盖子。表 14. DNA 连接的孵育条件
步 孵化温度 孵化时间 1 4°C 1分钟 2 20°C 15 分钟 3 4°C 抓住
4. 加入 50 µl 无核酸酶水,使每个样品达到 100 µl。
5. 加入 130 µl QIAseq Beads,然后涡旋混合。
6. 在室温下孵育 5 分钟。
7. 将管/板放在磁架上 2 分钟(用于管)或 10 分钟(用于板)。
8. 溶液澄清后,磁珠仍在磁架上,小心取出并丢弃上清液。
重要提示:不要丢弃磁珠,因为它们含有感兴趣的 DNA。
9. 加入 80 µl Nuclease-free Water 重悬磁珠,然后加入 128 µl QIAseq NGS Bead Binding Buffer。通过涡旋混合并在室温下孵育 5 分钟。
10. 将管/板放在磁架上 2 分钟(用于管)或 10 分钟(用于板)。溶液清除后,磁珠仍在磁架上,小心取出并丢弃上清液。
11. 磁珠仍在磁力架上,加入 200 µl 80% 乙醇。旋转管子 2-3 次或在磁铁的 2 个柱位置之间左右移动板以清洗珠子。小心取出并丢弃洗涤液。
12. 重复乙醇洗涤。
重要提示:在第二次洗涤后彻底清除所有乙醇痕迹。首先用 200 µl 移液器吸头去除乙醇,短暂旋转,然后使用 10 µl 移液器吸头去除任何残留的乙醇。
13. 磁珠仍在磁力架上,在室温下风干 5-10 分钟。
注意:目视检查颗粒以确认其完全干燥。如果颗粒没有完全干燥,可能需要额外的干燥时间。
14. 从磁力架上取下磁珠,然后加入 19 µl Nuclease-free Water 从磁珠上洗脱 DNA。通过移液充分混合。
15. 将管/板放回磁架,直到溶液清除。
16. 将 16.62 µl 上清液转移到清洁管/板上。
继续 ”操作步骤:逆转录”. 或者,样品可以在 -30 至 -15°C 的恒温冰箱中储存。 -
逆转录
开始前的要点
· 16.62 µl 产品来自“方案:DNA 连接”, 是该操作步骤的起始材料。
· 在冰上建立反应。
· 不要涡旋任何试剂或反应。
· QIAseq Beads 用于所有反应净化。
· 重要提示:每天准备新鲜的 80% 乙醇。
· 确保 QIAseq Beads 始终完全混合。这需要快速工作并在使用前立即重新悬浮珠子。如果操作步骤出现延迟,只需涡旋磁珠即可。
程序
1. 准备逆转录所需的试剂。
1a.解冻 Multimodal RT Primer;多模式 RT 缓冲液,5x;和 Multimodal RT Enhancer 在室温下。
1b.轻弹试管混合,然后短暂离心。
注意:RNase Inhibitor 和 EZ Reverse Transcriptase 应在使用前从冰箱中取出并置于冰上。使用后,立即将酶放回冰箱。
2. 根据准备逆转录组合表 15。短暂离心,上下吹打混合 10-12 次,然后再次短暂离心。
注意:如果设置了多个反应,请准备体积大于 10% 的预混液比反应总数所需的数量。表 15. 逆转录反应混合物
零件 体积/反应 样品(来自“方案:DNA 连接”) 16.62 µl 多模式 RT 引物 1µl 多模式 RT 缓冲液,5x 5 µl 多模式 RT 增强剂 0.5µl RNase 抑制剂 0.63µl EZ 逆转录酶 1.25µl 全部的 25µl
3. 根据以下步骤在热循环仪中孵育反应表 16。使用仪器的加热盖。表 16. 逆转录的孵育条件
步 孵化温度 孵化时间 1 4°C 1分钟 2 25°C 10 分钟 3 42°C 45 分钟 4 70°C 15 分钟 5 4°C 抓住
4. 添加 75 µl 无核酸酶水,使每个样品达到 100 µl。
5. 加入 130 µl QIAseq Beads 并通过涡旋或上下吹打数次混合。
6. 在室温下孵育 5 分钟。
7. 将管/板放在磁架上 2 分钟(用于管)或 10 分钟(用于板)。
8. 溶液澄清后,磁珠仍在磁架上,小心取出并丢弃上清液。
重要提示:不要丢弃磁珠,因为它们含有感兴趣的 DNA。
9. 磁珠仍在磁力架上,加入 200 µl 80% 乙醇。旋转管子 2-3 次或在磁铁的 2 个柱位置之间左右移动板以清洗珠子。小心取出并丢弃洗涤液。
10. 重复乙醇洗涤。
重要提示:第二次洗涤后,彻底清除所有乙醇痕迹。首先用 200 µl 移液器吸头除去乙醇,短暂旋转,然后使用 10 µl 移液器吸头除去任何残留的乙醇。
11. 磁珠仍在磁力架上,在室温下风干 5-10 分钟。
注意:目视检查颗粒以确认其完全干燥。如果颗粒没有完全干燥,可能需要额外的干燥时间。乙醇残留到下一个通用 PCR 步骤将影响 PCR 效率。
12.从磁力架上取下磁珠,然后加入 15 µl Nuclease-free Water 从磁珠上洗脱 DNA。
重要提示:如果执行分离的 DNA 和 RNA 靶标富集(附录 B),加入 22.4 µl Nuclease-free Water 洗脱。
13. 将管/板放回磁架,直到溶液清除。
14. 将 12.4 µl 上清液转移到干净的试管/板上。
重要提示:如果进行分离的 DNA 和 RNA 靶标富集(附录 B),将 10.2 µl 洗脱液转移到 2 个试管中的每个试管中,然后继续执行附录 B 操作步骤。
15. 继续 “方案:在单个试管中结合靶向 DNA+RNA 富集”. 或者,样品可以在 –30 至 –15°C 的温度下储存在恒温冰箱中。 -
在单个试管中结合靶向 DNA+RNA 富集
开始前的要点
· 12.4 µl 产品来自“操作步骤:逆转录”, 是该操作步骤的起始材料。
· 在冰上建立反应。
· 不要涡旋任何试剂或反应。
· 最终的文库双样本索引由 QIAseq Multimodal N7 Plate 和 QIAseq Multimodal S5 Plate 组合确定。 QIAseq Beads 用于所有反应净化。
重要提示:要使用此操作步骤,需要满足以下条件之一:
· QIAseq Multimodal Index I (12)(货号 333962)
· QIAseq Multimodal Index I Set A (96)(货号 333965)· QIAseq Multimodal Index I Set B (96)(货号 333975)
这些板有 12 或 48 反应形式,允许对 12 个 DNA 和 RNA 样品或 48 个 DNA 和 RNA 样品进行索引(使用一组 A 或 B 组的一个板)。在可切割板的每个指示孔中,都有用于 DNA 和 RNA 的干燥 N7 索引引物。板可以按列切割,以便对所需数量的样品进行索引。
每个索引试剂盒包括两个 A 组或 B 组板,用于制作总共 96 个 DNA 和 96 个 RNA 文库。通过组合 A 组和 B 组,可以复用多达 96 个 DNA 和 96 个 RNA 文库。板中的每个孔都是一次性使用。
· 重要提示:测序样品设置表中描述了所需的索引组合:
· 样品表多式联运 UDI 集 A:www.qiagen.com/PROM-15281
· 样品表多式联运 UDI 集 B:www.qiagen.com/PROM-15282
· 样品表多式联运 UDI 集 A 和集 B:www.qiagen.com/PROM-15283
· 重要提示:每天准备新鲜的 80% 乙醇。
· 确保 QIAseq Beads 始终完全混合。这需要快速工作并在使用前立即重新悬浮珠子。如果操作步骤出现延迟,只需涡旋磁珠即可。
程序
1. 准备目标富集所需的试剂。
1a.解冻 TEPCR 缓冲液,5x;多式联运 DHS 小组 (DNA);和多模式 VHS 面板 (RNA);并将 QIAseq Multimodal N7 Plate 置于室温。
1b.轻弹试管混合,然后短暂离心。
注意:QIAseq Multimodal N7 Plate 只需离心,无需混合。HotStarTaq DNA 聚合酶应在使用前从冰箱中取出并置于冰上。使用后,立即将酶放回冰箱。
2. 根据准备目标富集组合表 17。短暂离心,上下吹打混合 10-12 次,然后再次短暂离心。
注意:如果设置了多个反应,则准备的预混液体积比反应总数所需的量大 10%。表 17. 目标富集的反应混合物
零件 体积/反应 样品(来自“操作步骤:逆转录“) 12.4µl TEPCR 缓冲液,5x 8µl 多模态国土安全部组合(DNA) 10µl 多模态 VHS 面板(RNA) 8µl HotStarTaq DNA 聚合酶(6 U/μl) 1.6µl 全部的 40µl
3. 将 40 µl 目标富集反应混合物添加到 QIAseq Multimodal N7 板的孔中(表 18,表 19,和表 20),它们是可切割的板,在同一孔中包含用于 DNA 和 RNA 样品的预分配、干燥的索引引物对。
注意:板可以按列切割,以便对所需数量的样品进行索引。表 18. QIAseq Multimodal N7 板的布局,12 个反应
1 2 3 4 5 6 7 8 9 10 11 12 A DNAp-M001
S1
RNAp-M049
DNAp-M009
S9
RNAp-M057
Empty Empty Empty Empty Empty Empty Empty Empty Empty Empty B DNAp-M002
S2
RNAp-M050
DNAp-MOW
S10
RNAp-M058
Empty Empty Empty Empty Empty Empty Empty Empty Empty Empty C DNAp-M003
S3
RNAp-MO51
DNAp-MO11
S11
RNAp-M059
Empty Empty Empty Empty Empty Empty Empty Empty Empty Empty D DNAp-M004
S4
RNAp-M052
DNAp-M012
S12
RNAp-MOGO
Empty Empty Empty Empty Empty Empty Empty Empty Empty Empty E DNAp-M005
S5
RNAp-M053
Empty Empty Emply Empty Empty Empty Empty Empty Empty Empty Empty F DNAp-MQ06
S6
RNAp-M054
Empty Empty Empty Empty Empty Empty Empty Empty Empty Empty Empty G DNAp-M007
S7
RNAD-M055
Empty Empty Empty Empty Empty Empty Empty Empty Empt/ Empty Empty H DNAp-M008
S8
RNAp-M056
Empty Empty Empty Empty Empty Empty Empty Empty Empty Empty Empty
表 19. QIAseq Multimodal N7 板的布局,48 次反应 Set A1 2 3 4 5 6 7 8 9 10 11 12 A DNAp-MOOl
S1
RNAp-M049
DHAP-M009
S9
RNAp-M057
DNAp-M017
S17
RNAp-MD65
DNAp-M025
S25
RNAp-MO73
DNAp-M033
S33
RNAp-MO81
DNAp-M041
S41
RNAp-MOBS
Empty Empty Empty Empty Empty Empty B DNAp-M002
S2
RNAp M050
DNAp-M010
S10
RNAp M058
DNAp-M018
S18
RNAp M066
DNAp-MO26
S26
RNAp M074
RNAp-MO34
S34
RNAp M082
ONAp-M042
S42
RNAp M090
Empty Empty Empty Empty Empty Empty C DNAp-M003
S3
RNAp-M051
DNAp-MO11
S11
RNAp-M059
DNAp-MD19
S19
RNAp-M067
DNAp-MO27
S27
RNAp-M75
DNAp-M035
S35
RNAp-M083
DNAp-M043
S43
RNAp-M091
Empty Empty Empty Empty Empty Empty D DNAp-MOQ4
S4
RNAp-M052
DNAp-MO12
S12
RNAp-M060
DNAp-MD20
S20
RNAp-M068
DNAP-M02S
S28
RNAp-M076
DNAp-M036
S3
RNAp-M084
DNAp-M044
S44
RNAp-M092
Empty Empty Empty Empty Empty Empty E DNAp-M005
S5
RNAp - M053
DNAp-M013
S13
RNAP-M061
DNAp-MD21
S21
RNAp-M069
DNAp-MO2S
S29
RNAp-MO77
DNAp-MO37
S37
RNAp-M085
DNAp-MO45
S45
RNAp-M093
Empty Empty Empty Empty Empty Empty F DNAp-MOOG
S6
RNAp-M054
DNAp-MO14
S14
RNAp-M062
DNAp-M022
S22
RNAp-M070
DNAp-M030
S30
RNAp-M076
DNAp-M038
S38
RNAp - M086
DNAp-M046
S46
RNAp-M094
Empty Empty Empty Empty Empty Empty G DNAp M007
S7
RNAp-M055
DNAp M015
S15
RNAp-M063
DNAp M023
S23
RNAp-M071
DNAp M031
S31
RNAp-M07S
DNAp MO3g
S39
RNAp-M087
DNAp M047
S47
RNAp-MQ95
Empty Empty Empty Empty Empty Empty H DNAp-MOOB
S8
RNAp-M056
DNAp-M016
S16
RNAp-MD64
DNAp-M024
S24
RNAp-MD72
DNAp-M032
S32
RNAp-MO8fl
DNAp-M04C
S40
RNAp-M08S
DNAp-M048
S48
RNAp-M096
Empty Empty Empty Empty Empty Empty
表 20. QIAseq Multimodal N7 板的布局,48 次反应 Set B1 2 3 4 5 6 7 B 9 10 11 12 A DNAp-M097
S49
RNAp-M145
DNAp-M105
S57
RNAp-M153
DNAp-M113
S65
RNAp-M161
DNAp-M121
S73
RNAp-M169
DNAp-M129
S81
RNAp-M177
DNAp-M137
S89
RNAp-M185
Empty Empty Empty Empty Empty Empty B DNAp-M098
S50
RNAp-M146
DNAp-M106
S58
RNAp-M154
DNAp-M114
S66
RNAp-M162
DNAp-M122
S74
RNAp-M170
DNAp-M130
S82
RNAp-M178
DNAp-M138
S90
RNAp-M186
Empty Empty Empty Empty Empty Empty C DNAp-M099
S51
RNAp-Ml47
DNAp-M107
S59
RNAP-M155
DNAp-M115
S67
RNAp-M163
DNAp-M123
S75
RNAp-M171
DNAp-M131
S83
RNAp-M179
DNAp-M139
S91
RNAp-M187
Empty Empty Empty Empty Empty Empty D DNAp M100
S52
RNAp-M148
DNAp-M108
S60
RNAp-M156
DNAp M116
S88
RNAp-M164
DNAp-M124
S76
RNAp-M172
DNAp M132
S84
RNAp-M180
DNAp M14Q
S92
RNAp-M18fl
Empty Empty Empty Empty Empty Empty E DNAp-M101 S53 RNAp-M149 DNAp-M109 S61 RNAp-M157 DNAp-M117
S69
RNAp-M165
DNAp-M125 S77
RNAp-M173
DNAp-M133 S85 RNAp-M181 DNAp-M141
S93
RNAp-M189
Empty Empty Empty Empty Empty Empty F DNAp-M102
S54
RNAp-Ml50
DNAp-M110
S62
RNAp-M153
DNAp-M118
S70
RNAp-M166
DNAp-M126
S78
RNAp-M174
DNAp-M134
S86
RNAp-M182
DNAp-M142
S94
RNAp-M190
Empty Empty Empty Empty Em pa Empty G DNAp-M103
S55
RNAp-M151
DNAp-M111
S63
RNAp-M159
DNAp-M119
S71
RNAp-M167
DNAp-M127
S79
RNAp-M175
DNAp-M135
S37
RNAp-M133
DNAp-M143
S95
RNAp-M191
Empty Empty Empty Empty Empty Empty H DNAp-M104
S56
RNAp-M152
DNAp-M112
S64
RNAp-M160
DNAp-M120
S72
RNAp-M168
DNAp-M128
S80
RNAp-M176
DNAp-M136
S88
RNAp-M184
DNAp-M144
S96
RNAp-M192
Empty Empty Empty Empty Empty Empty
4. 短暂离心,上下吹打混合 8 次,然后再次短暂离心。
注意:如果只使用一个柱子,从可切割板上切下该柱子,然后继续下一步。
5. 使用中的循环条件对热循环仪进行编程表 21(DNA+RNA 引物<1500) 或表 22(DNA+RNA 引物≥1500)。表 21. 如果 DNA+RNA 引物 <1500,靶标富集的循环条件
步 时间 温度 初始变性 13 分钟 95°C 2 分钟 98°C 8个周期 15 秒 98°C 10 分钟 68°C 抓住 5分钟 72°C ∞ 4°C
表 22. 引物数≥1500/管时靶标富集的循环条件步 时间(1500–12,000 引物/管) 时间(>12,000 引物/管) 温度 初始变性 13 分钟 13 分钟 95°C 2 分钟 2 分钟 98°C 6个周期 15 s 15 s 98°C 15 分钟 30分钟 65°C 1个周期 5分钟 5分钟 72°C 抓住 5分钟 5分钟 4°C 抓住 ∞ ∞ 4°C
6. 将目标富集反应放入热循环仪并开始运行。
7. 运行完成后,添加 60 µl 无核酸酶水,使每个样品达到 100 µl。
8. 加入 100 µl QIAseq Beads 并通过涡旋或上下吹打数次混合。
9. 在室温下孵育 5 分钟。
10. 将管/板放在磁架上 2 分钟(用于管)或 10 分钟(用于板)。
11. 溶液澄清后,磁珠仍在磁架上,小心取出并丢弃上清液。
重要提示:不要丢弃磁珠,因为它们含有感兴趣的 DNA。
重要提示:当 DNA+RNA 引物≥12000 时,加入 75 µl Nuclease-free Water 重悬磁珠,然后加入 75 µl QIAseq Bead Binding Buffer。通过上下涡旋或移液混合。重复步骤 9 到 11。
12. 磁珠仍在磁力架上,加入 200 µl 80% 乙醇。旋转管子 2-3 次或在磁铁的 2 个柱位置之间左右移动板以清洗珠子。小心取出并丢弃洗涤液。
13. 重复乙醇洗涤。
重要提示:第二次洗涤后,彻底清除所有乙醇痕迹。首先用 200 µl 移液器吸头除去乙醇,短暂旋转,然后使用 10 µl 移液器吸头除去任何残留的乙醇。
14. 磁珠仍在磁力架上,在室温下风干 5-10 分钟或更长时间。
注意:目视检查颗粒以确认其完全干燥。乙醇残留到下一个通用 PCR 步骤将影响 PCR 效率。
15. 从磁力架上取下磁珠,然后加入 25 µl Nuclease-free Water 从磁珠上洗脱 DNA。通过移液充分混合。
16. 将管/板放回磁架,直到溶液清除。
17. 将 24 µl 上清液转移到干净的试管/板上。这将在接下来的 2 个操作步骤中使用。
18. 继续 ”方案:通用 PCR 循环的 qPCR 测定“. 或者,可以将样品储存在 –30 至 –15°C 的恒温冰箱中。 -
通用 PCR 循环的 qPCR 测定
开始前的要点
· 两微升产品来自“方案:结合靶向 DNA+RNA 单管富集“,或者 ”附录 B:分离的靶向 DNA 和 单独管中的 RNA 富集“,是每种反应混合物的起始材料。
· 重要提示:此过程需要 EvaGreen,20x 水溶液,并且必须从 Biotium 购买(货号 31000-T,31000)
· 在冰上建立反应。
· 不要涡旋任何试剂或反应。
程序
1. 准备 qPCR 所需的试剂。
1a.解冻 UPCR 缓冲液,5x; DNA qPCR AMP 组;和 RNA qPCR AMP。设置在室温。
1b.轻弹试管混合,然后短暂离心。
注意:HotStarTaq DNA 聚合酶应在使用前从冰箱中取出并置于冰上。使用后,立即将酶放回冰箱。
2. 根据准备 qPCR 反应表 23用于 DNA 文库或表 24用于 RNA 文库。短暂离心,上下吹打混合 10-12 次,然后再次短暂离心。
注意:如果设置了多个反应,请准备体积大于 10% 的预混液比反应总数所需的数量。表 23. DNA 文库 qPCR 的反应混合物
零件 体积/反应 样品(来自“方案:结合靶向 DNA+RNA 富集单管“) 2 µl 或者 DNA 样本(来自“附录 B:分离的靶向 DNA 和 RNA在单独的管中富集“) UPCR 缓冲液,5x 2 µl 无核酸酶水 4.1 µl DNA qPCR AMP 套件 1µl HotStarTaq DNA 聚合酶 (6 U/µl) 0.4µl EvaGreen,20x 水溶液* 0.5µl 全部的 10µl *必须从 Biotium 购买(货号 31000-T、31000)。
表 24. RNA 文库 qPCR 的反应混合物
零件 体积/反应 样品(来自“方案:结合靶向 DNA+RNA 富集单管“,页43) 2 µl 或者 RNA 样本(来自“附录 B:分离的靶向 DNA 和 RNA在单独的管中富集“,页74) UPCR 缓冲液,5x 2 µl 无核酸酶水 4.1 µl RNA qPCR AMP 套装 1µl HotStarTaq DNA 聚合酶 (6 U/µl) 0.4µl EvaGreen,20x 水溶液* 0.5µl 全部的 10µl *必须从 Biotium 购买(货号 31000-T、31000)。
3. 使用中的循环条件对 qPCR 仪器进行编程表 25。
注意:不需要熔解曲线。表 25. RNA 文库 qPCR 的反应混合物
步 时间 温度 抓住 13 分钟 95°C 2 分钟 98°C 2 步循环 变性 15 s 98°C 退火/延伸* 2 分钟 62°C 周期数 30 个周期 抓住 ∞ 4°C *执行荧光数据收集。
4. 反应后,确定 CT 值。基于 CT 值,通用 PCR 循环数定义为 CT(qPCR)+3,用于 DNA 和 RNA 文库。
例如,如果 DNA qPCR 为 CT=19,则为 DNA 通用 PCR 执行 22 个循环。如果 RNA qPCR 为 CT=15,则为 RNA 通用 PCR 执行 18 个循环。
替代方法:
运行完成后,观察“日志视图”中的扩增图并使用“自动基线”定义基线。使用扩增图的“对数视图”,确定扩增曲线达到其平台期的循环,并减少使用 2 个循环。例如,如果在 CT 为 18 时达到平台期,则 16 是所需的通用 PCR 扩增循环数。
5. 一旦确定了通用 PCR 的扩增循环,继续“操作步骤:通用 PCR”, -
通用 PCR
开始前的要点
· 九微升产品来自“方案:结合靶向 DNA+RNA 富集 单管“,或者 ”附录 B:分离的靶向 DNA 和 RNA 富集分离管“,是每种反应混合物的起始材料。
· 扩增所需的循环数在“操作步骤:qPCR 通用 PCR 循环的测定”,
· 在冰上建立反应。
· 不要涡旋任何试剂或反应。
· QIAseq Multimodal S5 Plates 采用 12 或 48 反应形式,可分别对 12 个 DNA 和 RNA 样品或 48 个 DNA 和 RNA 样品进行索引。 QIAseq Multimodal S5 Plates 中的每个孔都是一次性使用的。 SQDIB001 至 SQDIB048 和 SQDIB097 至 SQDIB144 与通用 DNA 引物混合用于 DNA 文库扩增。 SQDIB0049 至 SQDIB096 和 SQDIB0145 至 SQDIB192 与通用 RNA 引物混合用于 RNA 文库扩增。 S5引物预计与N7引物配对使用,SQDIB001与DNApM001配对,SQDIB002与DNAp-M002配对等; SQDIB049 与 RNAp-M049 配对,SQDIB050 与 RNAp-M050 配对等。板可按列切割,以便对所需数量的样品进行索引。
· 最终的文库双样本索引由 QIAseq Multimodal N7 Plate 和 QIAseq Multimodal S5 Plate 组合确定。
· 重要提示:测序样品设置表中描述了所需的索引组合:
·样品表多式联运 UDI 集 A:www.qiagen.com/PROM-15281
·样品表多式联运 UDI 集 B:www.qiagen.com/PROM-15282
·样品表多式联运 UDI 集 A 和集 B:www.qiagen.com/PROM-15283· QIAseq Beads 用于所有反应净化。
· 重要提示:每天准备新鲜的 80% 乙醇。
· 确保 QIAseq Beads 始终完全混合。这需要快速工作并在使用前立即重新悬浮珠子。如果操作步骤出现延迟,只需涡旋磁珠即可。
程序
1. 准备通用 PCR 所需的试剂。
1a.解冻 UPCR 缓冲液,5x,并将 QIAseq Multimodal S5 Plate 置于室温。
1b.轻弹试管混合,然后短暂离心。注意:HotStarTaq DNA 聚合酶应在使用前从冰箱中取出并置于冰上。使用后,立即将酶放回冰箱。
2. 根据准备通用 PCR表 26用于 DNA 文库或表 27用于 RNA 文库。短暂离心,上下吹打混合 10-12 次,然后再次短暂离心。
注意:如果设置了多个反应,请准备体积大于 10% 的预混液比反应总数所需的数量。表 26. DNA 文库组分通用 PCR 的反应混合物
零件 体积/反应 样品(来自“方案:结合靶向 DNA+RNA 富集单管“) 9 µl 或者 DNA 样本(来自“附录 B:分离的靶向 DNA 和 RNA在单独的管中富集“) UPCR 缓冲液,5x 5 µl 无核酸酶水 10µl HotStarTaq DNA 聚合酶 (6 U/µl) 1µl 全部的 25µl
表 27. RNA 文库组分通用 PCR 的反应混合物零件 体积/反应 样品(来自“方案:结合靶向 DNA+RNA 富集单管“,页43) 9 µl 或者 RNA 样本(来自“附录 B:分离的靶向 DNA 和 RNA在单独的管中富集“,页74) UPCR 缓冲液,5x 5 µl 无核酸酶水 10µl HotStarTaq DNA 聚合酶 (6U/µl) 1µl 全部的 25µl
3. 至 QIAseq Multimodal S5 板(表 28,表 29,或者表 30),将 25 µl 用于 DNA 文库通用 PCR 的反应混合物添加到 DNA 孔(第 1-6 列),并将 25 µl 用于 RNA 文库通用 PCR 的反应混合物添加到 QIAseq 的 RNA 孔(7-12 列)多式联运 S5 板。
注意:QIAseq Multimodal S5 板是可切割的板,在单独的孔中包含用于 DNA 和 RNA 样品的预分配、干燥的索引引物。 DNA引物和RNA引物预计成对使用:例如样品1应使用DNA引物SQDIB001和RNA引物SQDIB049,样品2应使用DNA引物SQDIB002和RNA引物SQDIB050等。
注意:如果只使用一个柱子,从可切割板上切下该柱子,然后继续下一步。表 28. QIAseq Multimodal S5 板的布局,12 个反应
1 2 3 4 5 6 7 8 9 10 11 12 A S1
SQDIB001
uDNA Pr
S9
SQDIB009
uDNA Pr
Empty Empty Empty Empty S1
SQDIB049
uRNA Pr
S9
SQDIB057
uRNA Pr
Empty Empty Empty Empty B S2
SQDIB002
uDNA Pr
S10
SQDIB010
uDNA Pr
Empty Empty Empty Empty S2
SQDIB050
uRNA Pr
S10
SQDIB058
uRNA Pr
Empty Empty Empty Empty C S3
SQDIB003
uDNA Pr
S11
SQDIB011
uDNA Pr
Empty Empty Empty Empty 3
SQDIB051
uRNA Pr
S11
SQDIB059
uRNA Pr
Empty Empty Empty Empty D S4
SQDIB004
uDNA Pr
S12
SQDIB012
uDNA Pr
Empty Empty Empty Empty S4
SQDIB052
uRNA Pr
S12
SQDIB060
uRNA Pr
Empty Empty Empty Empty E S5
SQDIB005
uDNA Pr
Empty Empty Empty Empty Empty S5
SQDIB0S3
uRNA Pr
Empty Empty Empty Empty Empty F S6
SQDIB006
uDNA Pr
Empty Empty Empty Empty Empty S6
SQDIB054
uRNA Pr
Empty Empty Empty Empty Empty G S7
SQDIB007
uDNA Pr
Empty Empty Empty Empty Empty S7
SQDIB055
uRNA Pr
Empty Empty Empty Empty Empty H S8
SQDIB008
uDNA Pr
Empty Empty Empty Empty Empty S8
SQDIB0S6
uRNA Pr
Empty Empty Empty Empty Empty
表 29. QIAseq Multimodal S5 板的布局,48 次反应 Set A1 2 3 4 5 6 7 8 9 10 11 12 A SQDIB001
S1
uDNA Pr
SQDIB009
S9
uDNA Pr
SQDIB017
S17
uONA Pr
SQDIB025
S25
uDNA Pr
SQDIB033
S33
uDNA Pr
SQDIB041
S41
uDNA Pr
SQDIB049
S1
uRNA Pr
SQDIB057
S9
uRNA Pr
SQDIB065
S17
URNA Pr
SQDIB073
S25
uRNA Pr
SQDIB081
S33
uRNA Pr
SQDIB089
S41
uRNA Pr
B SQDIB002
S2
uDNA Pr
SQDIB010
S10
uDNA Pr
SQDIB018
S18
uDNA Pr
SQDIB026
S26
uDNA Pr
SQDIB034
S34
uDNA Pr
SQD1B042
S42
uDNA Pr
SQDIB050
S2
uRNA Pr
SQDIB058
S10
uRNA Pr
SQDIB066
S18
uRNA Pr
SQDIB074
S26
URNA Pr
SQDIB082
S34
uRNA Pr
SQDIB090
S42
uRNA Pr
C SQDIB003
S3
uDNA Pr
SQDIB011
S11
uDNA Pr
SQDIB019
S19
uONA Pr
SQDIB027
S27
uDNA Pr
SQDIB035
S35
uDNA Pr
SQDIB043
S43
uDNA Pr
SQDIB051
S3
uRNA Pr
SQDIB059
S11
uRNA Pr
SQDIB067
S19
URNA Pr
SQDIB075
S27
uRNA Pr
SQDIB083
S35
uRNA Pr
SQDIB091
S43
uRNA Pr
D SQDIB004
S4
uDNA Pr
SQDIB012
S12
uDNA Pr
SQDIB020
S20
uDNA Pr
SQDIB028
S28
uDNA Pr
SQDIB036
S36
uDNA Pr
SQDIB044
S44
uDNA Pr
SQDIB052
S4
uRNA Pr
SQDIB060
S12
uRNA Pr
SQDIB068
S20
uRNA Pr
SQDIB076
S28
uRNA Pr
SGDIB084
S36
uRNA Pr
SQDIB092
S44
uRNA Pr
E SQDIB005
S5
uDNA Pr
SQDIB013
S13
uDNA Pr
SQDIB021
S21
uDNA Pr
SQDIB029
S29
uDNA Pr
SQDIB037
S37
uDNA Pr
SQDIB045
S45
uDNA Pr
SQDIB053
S5
uRNA Pr
SQDIB061
S13
uRNA Pr
SQDIB069
S21
uRNA Pr
SQDIB077
S29
uRNA Pr
SQDIB085
S37
uRNA Pr
SQDIB093
S45
uRNA Pr
F SQDIB006
S6
uDNA Pr
SQD1B014
S14
uDNA Pr
SQDIB022
S22
uDNA Pr
SQDIB030
S30
uDNA Pr
SQDIB038
S38
uDNA Pr
SQDIB046
S46
uDNA Pr
SQDIB054
S6
uRNA Pr
SQDIB062
S14
uRNA Pr
SQDIB070
S22
uRNA Pr
SQDIB078
S30
uRNA Pr
SQDIB086
S38
uRNA Pr
SQD1B094
S46
uRNA Pr
G SQDIB007
S7
uDNA Pr
SQDIB015
S15
uDNA Pr
SQDIB023
S23
uDNA Pr
SQDIB031
S31
uDNA Pr
SQDIB039
S39
uDNA Pr
SQDIB047
S47
uDNA Pr
SQDIB055
S7
uRNA Pr
SQDIB063
S15
uRNA Pr
SQDIB071
S23
uRNA Pr
SQDIB079
S31
uRNA Pr
SQDIB087
S39
uRNA Pr
SQDIB095
S47
uRNA Pr
H SQDIB008
S8
uDNA Pr
S16
SQDIB016
uDNA Pr
S24
SQDIB024
uDNA Pr
S32
SQDIB032
uDNA Pr
S40
SQDIB040
uDNA Pr
S48
SQDIB048
uDNA Pr
S8
SQDIB056
uRNA Pr
S16
SQDIB064
uRNA Pr
S24
SQDIB072
uRNA Pr
S32
SQDIB080
uRNA Pr
S40
SQDIB088
uRNA Pr
SQDIB096
S48
uRNA Pr
表 30. QIAseq Multimodal S5 板的布局,48 次反应 Set B1 2 3 4 5 6 7 8 9 10 11 12 A SQDIB097
S49
uDNA Pr
SQDIB105
S57
uDNA Pr
SQDIB113
S65
uDNA Pr
SQD1B121
S73
uDNA Pr
SQDIB129
S81
uDNA Pr
SQDIB137
S89
uDNA Pr
SQDIB145
S49
uRNAPr
SQDIB153
S57
uRNA Pr
SQDIB161
S65
uRNAPr
SQDIB169
S73
uRNA Pr
SQDIB177
S81
uRNA Pr
SQDIB18!
S89
uRNA Pr
B SQDIB098
S50
uDNA Pr
SQDIB106
S58
uDNAPr
SQDIB114
S66
uDNA Pr
SQDIB122
S74
uDNA Pr
SQDIB130
S82
uDNA Pr
SQDIB138
S90
uDNA Pr
SQDIB146
S50
uRNA Pr
SQDIB154
S58
uRNA Pr
SQDIB162
S66
uRNAPr
SQDIB170
S74
uRNA Pr
SQDIB178
S82
uRNA Pr
SQDIB186
S90
uRNA Pi
C SQDIB099
S51
uDNA Pr
SQDIB107
S59
uDNAPr
SQDIB115
S67
uDNA Pr
SQDIB123
S75
uDNA Pr
SQDIB131
S83
uDNA Pr
SQDIB139
S91
uDNA Pr
SQDIB147
S51
uRNA Pr
SQDIB155
S59
uRNA Pr
SQDIB163
S67
uRNA Pr
SQDIB17
S75
uRNA Pr
SQDIB179
S83
uRNA Pr
SQDIB187
S91
uRNA Pl
D SQDIB100
S52
uDNA Pr
SQDIB108
S60
uDNAPr
SQDIB116
S68
uDNA Pr
SQD1B124
S76
uDNA Pr
SQDIB132
S84
uDNA Pr
SQDIB140
S92
uDNA Pr
SQDIB148
S52
uRNA Pr
SQDIB156
S60
uRNA Pr
SQDIB164
S68
uRNAPr
SQDIB172
S76
uRNA Pr
SQDIB180
S84
uRNA Pr
SQDIB185
S92
uRNA Pi
E SQDIB101
S53
uDNA Pr
SQDIB109
S61
uDNA Pr
SQDIB117
S69
uDNA Pr
SQDIB125
S77
uDNA Pr
SQDIB133
S85
uDNA Pr
SQDIB141
S93
uDNA Pr
SQDIB149
S53
uRNA Pr
SQDIB157
S61
uRNA Pr
SQDIB16S
S69
uRNAPr
SQDIB173
S77
uRNA Pr
SQDIB181
S85
uRNA Pr
SQDIB18
S93
uRNA Pl
F SQDIB102
S54
uDNA Pr
SQDIB110
S62
uDNAPr
SQDIB118
S70
uDNA Pr
SQDIB126
S78
uDNA Pr
SQDIB134
S86
uDNA Pr
SQDIB142
S94
uDNA Pr
SQDIB150
S54
uRNA Pr
SQDIB158
S62
uRNA Pr
SQDIB166
S70
uRNA Pr
SQDIB174
S78
uRNA Pr
SQDIB182
S86
uRNA Pr
SQDIB19C
S94
uRNA Pl
G SQDIB103
S55
uDNA Pr
SQDIB111
S63
uDNAPr
SQDIB119
S71
uDNA Pr
SQDIB127
S79
uDNA Pr
SQDIB135
S87
uDNA Pr
SQDIB143
S95
uDNA Pr
SQDIB151
S55
uRNA Pr
SQDIB159
S63
uRNA Pr
SQDIB167
S71
uRNA Pr
SQDIB175
S79
uRNA Pr
SQDIB183
S87
uRNA Pr
SQDIB191
S95
uRNA Pt
H SQDIB104
S58
uDNA Pr
SQDIB112
S64
uDNAPr
SQDIB120
S72
uDNA Pr
SQD1B128
S80
uDNA Pr
SQDIB136
S88
uDNA Pr
SQDIB144
S96
uDNA Pr
SQDIB152
S56
uRNA Pr
SQDIB160
S64
uRNA Pr
SQDIB168
S72
uRNAPr
SQDIB176
S80
uRNA Pr
SQDIB184
S88
uRNA Pr
SQD1B192
S96
uRNA Pi
4. 使用中的循环条件对热循环仪进行编程表 31。表 31. 通用 PCR 的循环条件
步 时间 温度 抓住 13 分钟 95°C 2 分钟 98°C 2 步循环 变性 15 s 98°C 退火/延伸 2 分钟 62°C 循环数 基于 ”方案:qPCR 测定 通用 PCR 循环数“, 页51 抓住 ∞ 4°C
5. 反应完成后,加入 75 µl 无核酸酶水,使每个样品达到 100 µl。
6. 加入 100 µl QIAseq Beads,然后通过涡旋或上下吹打数次混合。
7. 在室温下孵育 5 分钟。
8. 将管/板放在磁架上 2 分钟(用于管)或 10 分钟(用于板)以将珠子与上清液分离。
9. 溶液澄清后,磁珠仍在磁架上,小心取出并丢弃上清液。
重要提示:不要丢弃磁珠,因为它们含有感兴趣的DNA。
10. 磁珠仍在磁力架上,加入 200 µl 80% 乙醇。旋转管子 2-3 次或在磁铁的 2 个柱位置之间左右移动板以清洗珠子。小心取出并丢弃洗涤液。
11. 重复乙醇洗涤。
重要提示:第二次洗涤后,彻底清除所有乙醇痕迹。首先用 200 µl 移液器吸头除去乙醇,短暂旋转,然后使用 10 µl 移液器吸头除去任何残留的乙醇。
12. 磁珠仍在磁力架上,在室温下风干 5-10 分钟。
注意:目视检查颗粒以确认其完全干燥。如果颗粒没有完全干燥,可能需要额外的干燥时间。
13. 从磁力架上取下磁珠,然后加入 20 µl Nuclease-free Water 从磁珠上洗脱 DNA。通过移液充分混合。
14. 将管/板放回磁架,直到溶液清除。
15. 将 18 µl 上清液转移到干净的试管/板上。
16. 继续 ”建议:文库 QC 和量化“. 或者,可以将样品储存在 –30 至 –15°C 的恒温冰箱中。 -
文库 QC 和量化
NGS 文库质控
QC 可以使用安捷伦生物分析仪或 TapeStation 进行。检查文库片段的大小分布是否正确(约 400–500 bp 中值大小)以及是否缺少接头或接头二聚体(约 <200 bp)(图4).
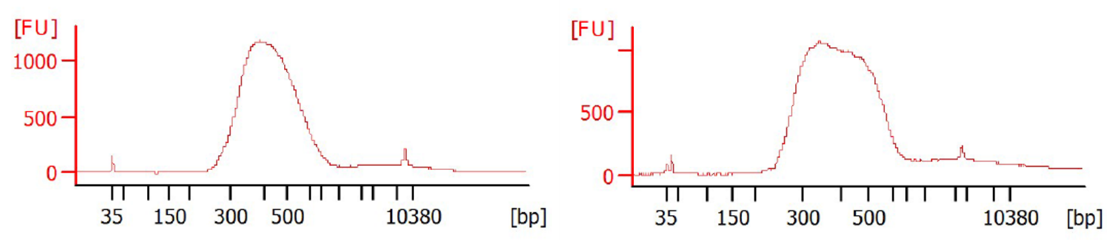
图 4. QIAseq 多模式靶向 DNA(左)和靶向 RNA(右)文库。△点击放大图片
首选文库量化方法来自 Bioanalyzer 或 TapeStation®的文库产量测量依赖于嵌入 DNA 或 RNA 的荧光染料。这些染料无法区分带有或不带有接头序列的分子,但只有具有完整接头序列的完整 QIAseq Multimodal 文库才会被测序。因此,强烈推荐使用 QIAGEN 的 QIAseq Library Quant Array Kit(货号 333304)或 QIAseq Library Quant Assay Kit(货号 333314),其中包含实验室验证的正向和反向引物以及 DNA 标准品用于对准备好的 QIAseq Multimodal 文库进行准确量化。
-
Illumina MiSeq 和 NextSeq 上的测序设置
开始前的要点
· 关于文库稀释浓度和文库加载浓度的建议基于 QIAseq Library Quant System。
· 在 Illumina 平台上进行测序时,必须使用 QIAseq A Read 1 Primer I(Custom Read 1 Sequencing Primer)和 Multimodal Read 2 Primer(Custom Read 2 Sequencing Primer)。
· QIAseq A Read 1 Primer I(Custom Read 1 Sequencing Primer)进入以下特定试剂盒位置:
· MiSeq 位置 #18
· NextSeq 位置 #7
· Multimodal Read 2 Primer(Custom Read 2 Sequencing Primer)进入以下特定试剂盒位置:
· MiSeq 位置 #20
· NextSeq 位置 #8
· 双端测序应用于 Illumina 平台上的 QIAseq Multimodal 文库。
· 读取 1:149 bp
· 读取 2:149 bp
· 自定义索引 1:10 个基点
· 自定义索引 2:10 个基点
· 有关如何使测序文库变性、准备自定义索引引物和设置测序运行的完整说明,请参阅系统特定的 Illumina 文档。
· 包括特定于仪器的图像以帮助进行测序准备。
MiSeq 的测序准备工作1.从 QIAseq Multimodal Panel 的“资源”选项卡下载适当的模板:
· 样品表 Multimodal UDI Set A(也用于索引 1-12):www.qiagen.com/PROM-15281
· 样品表多式联运 UDI 集 B:www.qiagen.com/PROM-15282
· 样品表多式联运 UDI 集 A 和集 B:www.qiagen.com/PROM-15283
2.在模板上:
2a.修改 Investigator Name、Date、Sample_ID 和 Sample Name。
重要提示:我们建议在 DNA 文库的样本名称中添加 -DNA,在 RNA 文库中添加 -RNA,以便在数据分析期间自动解析 DNA 和 RNA 文库。如果文库没有标记,则必须手动将它们解析到 DNA 或 RNA 盒中。
2b.删除任何未使用的索引对并保存样本表以供上传。2c.Read 1 为 149 bp,Read 2 为 149 bp,每个 Index Read 为 10 bp。
3. 样品稀释和汇集:将文库稀释至 2 或 4 nM,用于 MiSeq。然后,如果每个文库需要相似的测序深度,则以等摩尔量组合具有不同样本索引的文库。
注意:如果将具有相同数量引物的库组合在一起,则将等量的单个库以 4 nM 汇集在一起。如果文库有不同的引物编号,则根据它们的引物数量以体积比组合文库。例如,文库 A 有 5000 个 4 nM 的引物,文库 B 有 600 个 4 nM 的引物;将 50 µl 文库 A 与 6 µl 文库 B 结合起来,在同一次测序运行中,文库 A 和 B 的覆盖深度相似。
4. 文库准备和加载:根据 MiSeq 系统变性和稀释文库指南在 MiSeq 上准备和加载文库。 MiSeq 上的最终库浓度为 10-12 pM。
注意:关于文库加载浓度的建议基于 QIAseq Library Quant System。
(杂交缓冲液)从 Illumina Sequencing Kit 中稀释 3 µl QIAseq A Read 1 Primer I(已提供)以获得 0.5 µM 的最终浓度。将 600 µl 稀释的 QIAseq A Read 1 Primer I 加载到 MiSeq 试剂盒的位置 18(图5).有关更多详细信息,请参阅 Illumina 的 MiSeq 系统:自定义引物指南。
5. 用于读取 2 制备和加载的定制测序引物:使用 Illumina 测序试剂盒中的 597 µl HT1(杂交缓冲液)稀释 3 µl Multimodal Read 2 Primer(已提供),以获得 0.5 µM 的最终浓度。将 600 µl 稀释的 QIAseq Read 2 Primer 加载到 MiSeq 试剂盒的位置 20(图5).有关更多详细信息,请参阅 Illumina 的 MiSeq 系统:自定义引物指南。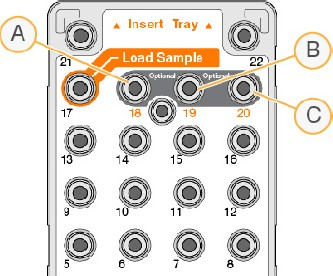
图 5. MiSeq 试剂盒。 A:Read 1 Custom Primer 的位置 18; C:Read 2 Custom Primer 的位置 20。△点击放大图片
6. 完成测序运行后,继续“操作步骤:数据分析使用 QIAseq 多模态数据分析门户“.
NextSeq 的测序准备工作1. 样品稀释和汇集:将文库稀释至 0.5、1、2 或 4 nM,用于 NextSeq。然后,如果每个文库需要相似的测序深度,则以等摩尔量组合具有不同样本索引的文库。
注意:文库稀释浓度建议基于 QIAseq Library Quant System。
注意:如果将具有相同数量引物的库组合在一起,则将等量的单个库以 4 nM 汇集在一起。如果文库有不同的引物编号,则根据它们的引物数量以体积比组合文库。例如,文库 A 有 5000 个 4 nM 的引物,文库 B 有 600 个 4 nM 的引物;将 50 µl 文库 A 与 6 µl 文库 B 结合起来,在同一次测序运行中,文库 A 和 B 的覆盖深度相似。
2. 文库准备和加载:根据 NextSeq 系统变性和稀释文库指南准备文库并将其加载到 NextSeq。 NextSeq 上的最终文库浓度为 1.2–1.5 pM。
注意:关于文库加载浓度的建议基于 QIAseq Library Quant System。
3. 用于 Read 1 制备和加载的定制测序引物:使用 Illumina 测序试剂盒中的 1994 µl HT1(杂交缓冲液)稀释 6 µl QIAseq A Read 1 Primer I(已提供),以获得 0.3 µM 的最终浓度。将 2 ml 稀释的 QIAseq A Read 1 Primer I 加载到 NextSeq 试剂盒的位置 7(图6).注意:所有其他步骤均参考 NextSeq 500 系统指南(部件号 15046563)或 NextSeq 550 系统指南(部件号 15069765-02)中描述的运行设置工作流程。
Illumina Sequencing Kit 中的(杂交缓冲液)稀释 6 µl Multimodal Read 2 Primer 以获得 0.3 µM 的最终浓度。将 2 ml 稀释的 Multimodal Read 2 Primer 加载到 NextSeq 试剂盒的位置 8(图6).
图 6. NextSeq 试剂盒。△点击放大图片
4. 使用 QIAseq Multimodal 定制 UDI 时,使用仪器上的 Local Run Manager (LRM) V2 上传样品表(请参阅第 66 页下载适当的模板和修改模板)并继续测序:读取 1 为 149 bp,读取2 为 149 bp,每个 Index Read 为 10 bp。
5. 完成后,继续“操作步骤:使用 QIAseq Multimodal 进行数据分析 数据分析门户“. -
使用 QIAseq 多模式数据分析门户进行数据分析
测序后,可以使用 QIAGEN 的 QIAseq Multimodal Data Analysis Portal 分析结果。我们的数据分析管道将执行到参考基因组的映射、UMI 计数、读取修剪(去除引物序列)和变异识别。所有检测到的变体都可以使用 QCI-I 进一步解释。
1. 转到 QIAseq 多模式数据分析门户,ngsdataanalysis2.qiagen.com/MultiModal/
2. 登录门户。
3. 在读取文件选项卡中,选择 BaseSpace 以从 BaseSpace 上传文件,或选择已上传 > 上传新文件以从本地硬盘上传文件。
△点击放大图片
注意:已上传到门户的所有文件都列在“读取文件”选项卡下。在这里可以删除不再需要的文件并与协作者共享文件。
4. 选择要分析的文件旁边的框,然后单击选择分析。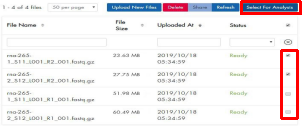
△点击放大图片
5. 在分析作业选项卡下,按照下拉菜单和复选框配置分析,如下所述: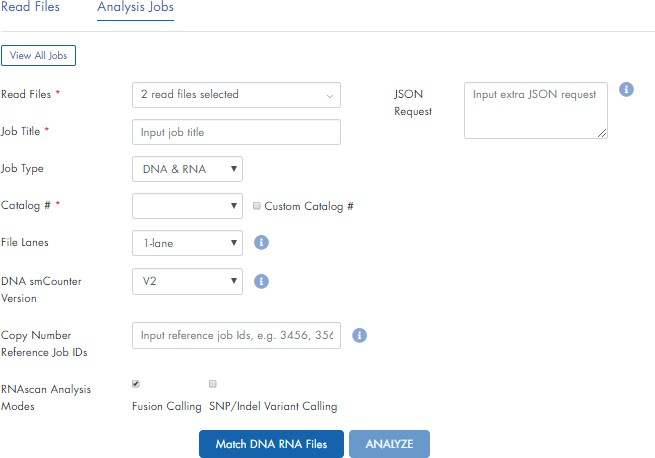
△点击放大图片
· 读取文件:验证是否选择了正确的读取文件。
· 工作标题:输入分析工作的标题。
· 工作类型:选择 DNA 和 RNA、仅 DNA 或仅 RNA。
· 目录编号:如果使用目录面板,请从下拉菜单中选择编号。如果使用自定义面板,请手动输入自定义目录号。
· 文件通道:如果使用 MiSeq/HiSeq/NovaSeq 或 NextSeq 与级联文件一起使用,则选择 1 通道。如果使用 NextSeq 而没有最终连接文件,则选择 4-lane。
· DNA smCounter版本:根据实验选择合适的版本需要。如需指导,请选择信息。
· 拷贝数参考作业 ID:对于拷贝数分析,需要在设置案例样本之前使用门户网站分析正常样本。输入与您的对照样本对应的作业 ID 以进行拷贝数分析。
· RNAscan 分析模式:选择 Fusion Calling 和/或 SNP/Indel Variant Calling。默认情况下,每个选择都提供基因表达分析。
6. 单击匹配 DNA RNA 文件以手动将选定的读取文件拖到 DNA 文件框或 RNA 文件框。
注意:我们建议您在 DNA 文库的样本名称末尾包含 -DNA,在 RNA 文库中包含 -RNA,以便在数据分析期间自动解析 DNA 和 RNA 文库。
7. 单击分析。分析作业状态从“Queued”变为“In progress”,然后变为“Done successful”。
8. 分析完成后,可以通过单击下载来下载输出文件。注意:最终,检测到的变异可以用 QCI-I 解释。


